個人による個人のための
バイオを実践する3日間
2019年3月1日〜3日、バイオテクノロジーの基礎と応用可能性を学ぶ3日間の集中ワークショップ「YCAM InterLab Camp vol.3」が開催された。
「YCAM InterLab Camp」は、さまざまな領域で活躍する研究者やアーティストを迎え、メディアアートに関連する技術やツールについて学ぶワークショップ・シリーズだ。2008年のスタート以来、第一弾として「openFrameworks」、第二弾には「TECHTILE(テクタイル)」を取り上げるなど、野心的なテーマに挑んできた。
そんな YCAM InterLab Camp が第三弾のテーマに選んだのは、「パーソナル・バイオテクノロジー」だ。その背景には、近年急速な盛り上がりを見せる「DIYバイオ」ムーブメントがある。かつて企業や研究機関のラボの中でしか行えなかったバイオテクノロジーは、デバイスの進化やDNA解析・合成コストの低下により、一般市民が扱える研究領域へと開放されつつある。
YCAMでは、こうした流れにいち早く呼応し、2015年度にバイオラボを開設。市民向けのバイオ関連イベントを多数開催してきた。今回のワークショップは、今まさに揺籃期を迎えようとする「個人による個人のためのバイオ=パーソナル・バイオテクノロジー」を実践する試みであり、YCAMがこれまで積み上げてきた知見の集大成といえるだろう。
今回は3日間にわたり、DNAに関わる技術を活用した生命情報の「読み」「書き」のプロセスを学んでいく。さらにバイオテクノロジーをめぐる「生命倫理」についても議論を深め、応用可能性を探求していく密度の濃い内容だ。
講師には、それぞれの領域における国内外のトップランナーたちを招聘。多彩なレクチャーを通じて、バイオテクノロジーに関する世界動向や最新の知見に触れる貴重な機会となった。

1オリエンテーション&エクササイズYCAM Bio Research, contact Gonzo
社会実装が進むバイオを
みんなで学び、考える
ワークショップ1日目は、YCAM R&Dディレクター・伊藤隆之氏によるレクチャーからスタートした。ふだんはシアター・スペースとして使われているスタジオAに集まった参加者たちは、やや緊張した面持ちで話に聞き入った。
 YCAM R&Dディレクター・伊藤隆之氏
YCAM R&Dディレクター・伊藤隆之氏
「コンピュータがパーソナルになったように、バイオテクノロジーも今後、どんどんパーソナルなものになっていく。そう考えたことが、今回の企画につながっています」
企画立案の背景について語った伊藤氏は、その考えに至った理由の一つとして、バイオテクノロジーの主要な技術のひとつであるDNA解析コストの低下を挙げた。
「例えば、DNAを読み取るシーケンサーの性能が飛躍的に向上し、ヒト1人分のゲノム情報を解析するコストは、この15年ほどの間におよそ10万分の1まで低下しました。それに関わるプロセスで使用されるデバイス類も、DIYで制作するための設計図が公開されてきていたり、小型で安価なものが販売されるようになってきています。また、併行してバイオに関心のある市民やアーティストたちが次々にバイオハッカースペースを立ち上げ、バイオテクノロジーを使った革新的なプロジェクトに取り組むようになってきているのです」

このようなバイオの民主化が進行すれば、倫理的な問題を含め、市民にもバイオに関する一定の知識や理解が必要になってくる。そこで伊藤氏は、YCAMのメンバーが自らバイオを学び、その応用可能性を模索するフレームワークとして「YCAMバイオ・リサーチ」を立ち上げた。さらに館内にバイオラボを開設し、多彩なワークショップやフィールドワークを展開。こうした活動を通じて地域や市民を巻き込みながら、バイオに関するリテラシーの醸成のを目指しているという。
「ますます身近になるバイオを、みんなで学んで考える場にしていきたい」。そう語った伊藤氏は、今回のワークショップを「パーソナル・バイオテクノロジー時代の新たな学びのかたち」と位置付け、講演を締めくくった。
バイオに関心を持つ
多様な人々が国内外から参加
続いて、大阪を拠点に活動するアーティスト集団「contact Gonzo(コンタクト・ゴンゾ)」により、チームビルディングを兼ねたエクササイズが行われた。
 「contact Gonzo」の塚原悠也氏
「contact Gonzo」の塚原悠也氏
殴り合いとコンテンポラリーダンスの間で身体をぶつけ合うハードなパフォーマンスで知られる彼らだが、今回は木の枝を使って参加者同士を接続するワークを展開。枝でつながりあった接点の感覚は、互いの存在を強く認識させ、参加者たちの間にノンバーバルな対話を生んでいく。非常にシンプルなエクササイズながら、接触がもたらす情報のフィードバックによって思考が拡張される不思議な感覚を覚えた。
 長さも太さもバラバラな木の枝を使い、できるだけ多くの接点で互いをつなぎあう。
長さも太さもバラバラな木の枝を使い、できるだけ多くの接点で互いをつなぎあう。
場の空気が温まったところで、自己紹介タイムへ。今回、公募により国内外から集まった参加者は総勢34名。研究者やアーティストをはじめ、デザイナー、エンジニア、大学受験を終えたばかりの高校生など、バイオテクノロジーに関心を持つ多様な人々が参加していた。
 日本人男性初のインド政府認定アーユルヴェーダ医という小峰博生さん。「DIYバイオによって伝統医療の科学的根拠を探りたい」と話した。
日本人男性初のインド政府認定アーユルヴェーダ医という小峰博生さん。「DIYバイオによって伝統医療の科学的根拠を探りたい」と話した。 早稲田大学基幹理工学部准教授の橋田朋子さん。材料や制御に自然エネルギーを活用したマニュファクチャリングなどの研究をしている。
早稲田大学基幹理工学部准教授の橋田朋子さん。材料や制御に自然エネルギーを活用したマニュファクチャリングなどの研究をしている。 デザインエンジニアリングファーム「Takram」の伊東実さん。パーソナル・バイオテクノロジーやYCAMのコミュニティに関心があり、参加を決めたという。
デザインエンジニアリングファーム「Takram」の伊東実さん。パーソナル・バイオテクノロジーやYCAMのコミュニティに関心があり、参加を決めたという。
一部の経験者を除き、大半はプラクティカルな経験がないビギナーが占めていた。それぞれの肩書きや年齢、国籍はバラエティに富んでいたが、日々進化するバイオの技術だけではなく、その先に広がる可能性を自ら探求したいと意欲を述べる参加者の多さが印象的だった。
2昼食ゲノム弁当
ゲノミクスの進化を
味覚で体感する「ゲノム弁当」
ここで午前の部が終了し、ランチタイムへ。希望者に対して、YCAMと山口市内の料理店「ベジタブル喫茶 ToyToy」がコラボレーションした「ゲノム弁当」が提供された。
ゲノム弁当とは、ゲノム配列が決定済みの生物種だけを食材に使った弁当を開発するプロジェクトだ。例えばゲノム弁当が始まった2016年までにゲノムが解読された生物種は9000種以上に及ぶ。年々その数は増えているが、そういった生物種から100種ほどを食材に選び、メニューを構成している。
 この日のメニューは、ねぎのオリーブオイル煮、むらさきキャベツのしば漬け、10種以上のスパイスを使ったキーマカレーなどを詰め合わせた内容。
この日のメニューは、ねぎのオリーブオイル煮、むらさきキャベツのしば漬け、10種以上のスパイスを使ったキーマカレーなどを詰め合わせた内容。
ただし、この日提供されたのは「ゲノム無視スパイシー弁当」。文字通り、ゲノムとは無関係のふつうに美味しいお弁当だ。制作を担当した「ベジタブル喫茶 ToyToy」の村田敦氏は、その意図について「食べ方にグラデーションをつけたかった」と説明する。

「現状、風味付けに欠かせないスパイス類はほとんどがゲノム未解読。多彩な味付けを実現するには限界があります。ならば、ゲノム食材の世界を味覚の変化で体感してもらおうと、初日はあえてゲノムを無視したメニューにしようと考えました」
村田氏によれば「明日からはゲノム決定済み食材を使ったお弁当が出る」とのこと。さらに「3日目はオードブル形式で提供される」との予告もあり、その内容に期待が高まった。
 参加者たちは一品一品の味を確かめながら感想を述べ合い、それぞれ交流を楽しんでいる様子だった。
参加者たちは一品一品の味を確かめながら感想を述べ合い、それぞれ交流を楽しんでいる様子だった。
3実験DNA抽出、PCR
DNAという物質を情報として読む
午後は、いよいよ「DNAを読む」工程に入る。参加者は6つのテーブルに分かれ、2人1組で実験を進めていく。
 卓上には、DNA抽出に必要なさまざまな機材や試薬があらかじめ用意されていた。
卓上には、DNA抽出に必要なさまざまな機材や試薬があらかじめ用意されていた。
DNAを読むためのプロトコルについては、YCAM研究員でYCAMバイオ・リサーチの主要メンバーである津田和俊氏が説明を行なった。
 YCAM研究員の津田和俊氏
YCAM研究員の津田和俊氏
「DNA は、A(アデニン)、T(チミン)、C(シトシン)、G(グアニン)という4種類の塩基がペアを組んで二重らせん状に連なったもので、細胞核の中の染色体に折りたたまれて格納されています。塩基のつながり方には規則性があり、AはTと、GはCと必ずペアになります。この性質は「塩基の相補性」と呼ばれます。ゲノムとは、このDNAに記録されたすべての遺伝情報のことで、ヒトの場合、その情報量は約30億塩基(約3Gb)に相当します」
 細胞核の中にあるDNAは、ゲノムDNAと呼ばれる。一方、細胞の中には、ゲノムDNAだけでなく、ミトコンドリアや葉緑体がそれぞれに持つ固有のDNA(オルガネラDNA)も存在しているという。
細胞核の中にあるDNAは、ゲノムDNAと呼ばれる。一方、細胞の中には、ゲノムDNAだけでなく、ミトコンドリアや葉緑体がそれぞれに持つ固有のDNA(オルガネラDNA)も存在しているという。
「細胞の体内では、こうしたDNAの情報がRNAにコピーされ(転写)、そのRNA情報を基にタンパク質がつくられています(翻訳)。つまり、DNAは物質であると同時に情報でもあるわけです。物質としてのDNAから、情報である塩基配列データを読み取る。これが今日みなさんが取り組むDNAの“読み”の作業です」
津田氏から示されたDNAを読むための作業工程は、おおむね次のとおりだ。

今回、DNA抽出のために用意されたサンプルは、野菜14種類と料理9種類。そう、ランチに食べたゲノム弁当に使われていた食材とおかずである。「自分が実際に食べたもののDNAを読む」となると、遠い存在だったDNA解析が一気に身近なものに感じられる。

計23種のサンプルは下記の通り。大きく3パターンに分類される。
① 素材(全ゲノム配列決定済み、6種類):
トマト、人参、白菜、キャベツ、黒米、ひよこ豆
② 素材(全ゲノム配列未決定、8種類):
しめじ、大根、かぶ、じゃが芋、玉ねぎ、レンコン、ほうれん草、キヌア
③ 料理(9種類):
れんこんひよこ豆味噌煮(れんこん、ひよこ豆、ひよこ豆味噌、のり)
炊き込みごはんの具(大根、にんじん、しめじ、ひえしょうゆ、ごま油)
コロッケの中身(さつまいも、ひよこ豆味噌)
漬物 甘酢漬け(白菜、大根、水菜、酢(米)、砂糖(てんさい))
きのこスープ
ゲノムソース(味噌、アーモンド、しょうゆ、唐辛子、砂糖、ごま)
きのこ豆腐ハンバーグ タネ
オムレツ(玉子、じゃが芋、玉ねぎ、豆乳、オリーブ油)
人参サラダ(人参、キヌア、オリーブオイル、マスタードシード、くるみ、高菜)
①の素材については、すでにゲノムDNAの全配列が完全に決定されているため、公開されている解読済みデータ(リファレンス配列)に沿ってゲノムを解析することができる。一方、②の素材については、全ゲノムDNAについて概要のみが判明している不完全な状態のため、このデータ(ドラフト配列)を参照して解析するには膨大な時間が必要になる。③については、複数の異なる素材が混在しているので、それぞれのゲノムを正確に読み取ることがそもそも難しいという。
そこで今回は、②と③については、ゲノムDNAではなく葉緑体DNAの一部を読み、DNAバーコーディングと呼ばれる手法を使って生物種の同定を試みるという。
DNAバーコーディングとは、生物の種を特定しやすいDNA領域を生物種ごとに選び出してデータベース化し、バーコードのように参照することで、未知の生物種を特定する技術だ。植物においては、葉緑体DNAの中のmatK遺伝子や光合成に関わる rbcL遺伝子(約600塩基)が標準的なバーコード領域として利用されることが多い。
つまり、データベースに登録されている多様な rbcL遺伝子の配列と、今回の食材サンプルから抽出したDNAのrbcL遺伝子の領域の情報を突き合わせれば、植物の種の同定ができるわけだ。
説明を受けた後、各チームは中央のテーブルに移動し、それぞれサンプルを選んだ。しかし、参加者たちは「全ゲノム配列決定済み=ゲノムを解析しやすい」と考えたのか、ほとんどのチームが①の素材群からサンプルを選んでいた。ちなみに、出遅れた筆者のチームは「にんじんサラダ」で解析に挑むことになった。
身近な食材サンプルからDNAを抽出する
まずはゴム手袋をはめ、選んだサンプルをハサミで細かく刻んでいく。
 韓国から参加したアーティスト、セオ・ヒョジュンさんと、フランスから参加した高校生、赤枝ルカさんのペア。「食材をハサミで切るのは初めて」と笑う。
韓国から参加したアーティスト、セオ・ヒョジュンさんと、フランスから参加した高校生、赤枝ルカさんのペア。「食材をハサミで切るのは初めて」と笑う。
刻んだサンプルは、凍結乾燥の状態にするためにドライアイス(液体窒素の代替)を加えながら、乳鉢と乳棒で粉末状にすりつぶしていく。これが意外と力の要る作業で、なかなか粉末状にならない。乳鉢をしっかり固定していないと、中身が飛び出すこともある。
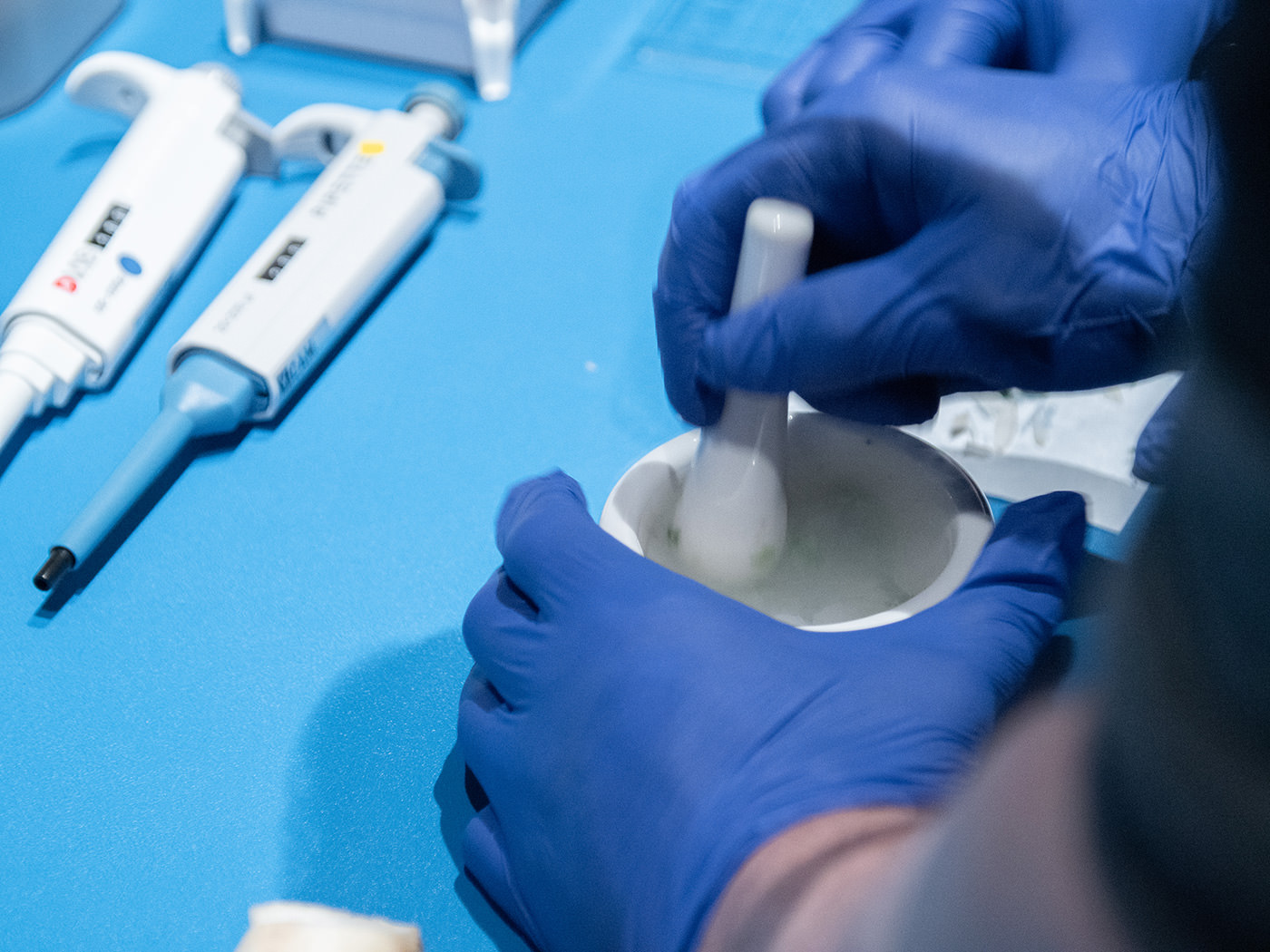 ドライアイスと混ぜ合わせていくと、ふりかけ状になっていく。
ドライアイスと混ぜ合わせていくと、ふりかけ状になっていく。 すりつぶしたサンプルは、用意されたマイクロチューブに移す。
すりつぶしたサンプルは、用意されたマイクロチューブに移す。 チューブスタンドは、YCAMがワークショップ用に開発したもの。スタッフがアクリル板をレーザーカッターで切り出してつくったという。工程が進むたびに、すごろくの駒のようにチューブを右へ移動させていく。
チューブスタンドは、YCAMがワークショップ用に開発したもの。スタッフがアクリル板をレーザーカッターで切り出してつくったという。工程が進むたびに、すごろくの駒のようにチューブを右へ移動させていく。
本来、DNA抽出の方法は、サンプルの種類によって異なるそうだ。そのため研究の現場では、実験と検証を何度も繰り返し、試薬成分の最適な構成を決めていくのだという。ただし今回は初心者に配慮し、市販の植物用DNA精製キットを使用。試薬もあらかじめ調合済みのものを使い、実験を行なった。
また、試薬やチューブの蓋にはサンプルの取り違えを防ぐチーム識別用のアルファベットが付記されており、主催側のこまやかな配慮が感じられた。
 各テーブルにはYCAMスタッフが1名づつついて、作業手順について的確に指示してくれる。サポート体制も万全だ。
各テーブルにはYCAMスタッフが1名づつついて、作業手順について的確に指示してくれる。サポート体制も万全だ。 伊藤氏も会場内をせわしなく移動しながら、各チームのサポートに奔走する。
伊藤氏も会場内をせわしなく移動しながら、各チームのサポートに奔走する。
続いて、ヒートブロックという機材でマイクロチューブを温め、ドライアイスを気化させる。ヒートブロックは熱処理や酵素処理に使用されるという。
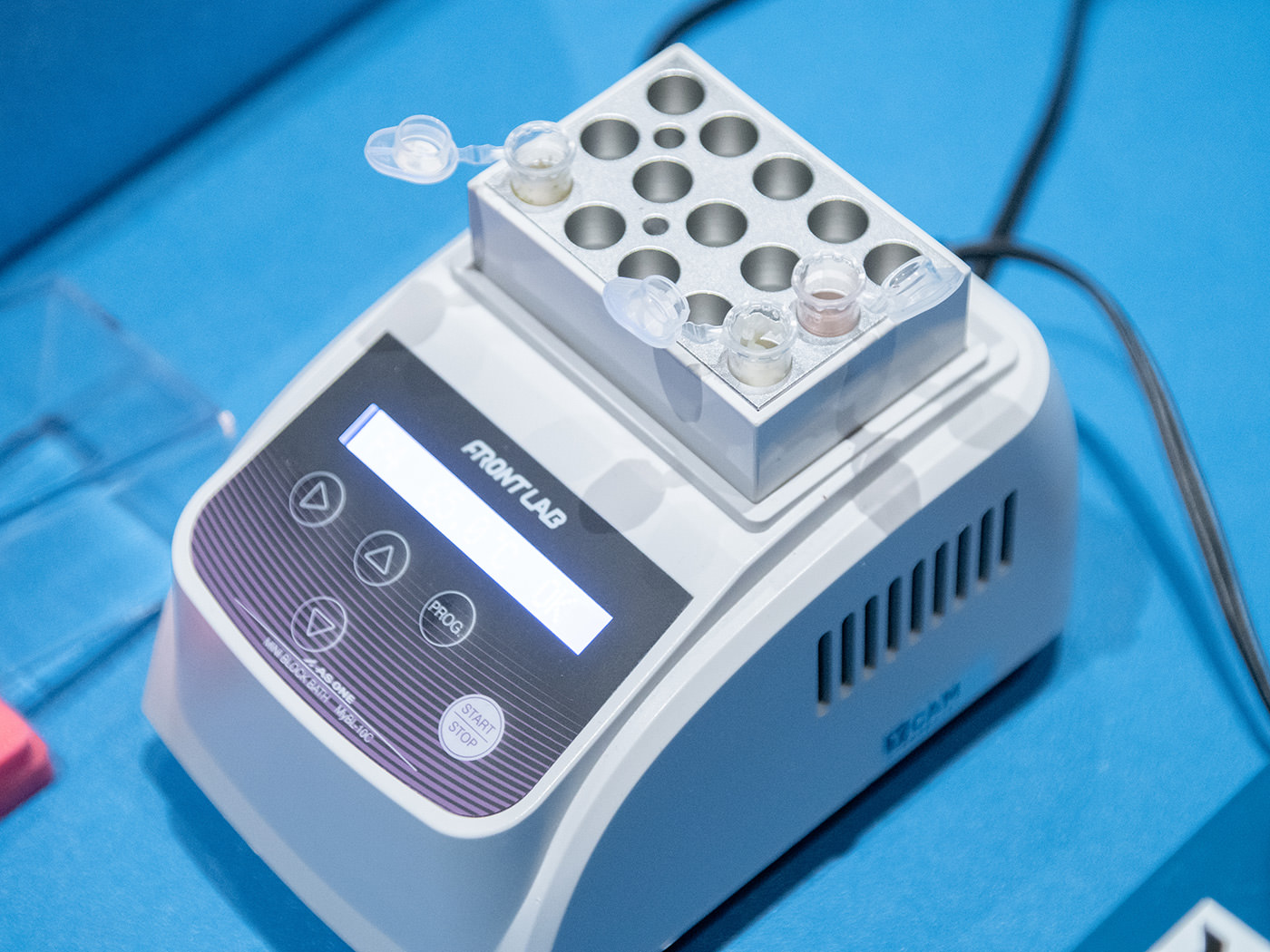 ドライアイスが気化し、蓋が開いてサンプルが飛び散るの破裂を防ぐため、蓋は開けたまま。
ドライアイスが気化し、蓋が開いてサンプルが飛び散るの破裂を防ぐため、蓋は開けたまま。
ドライアイスの気化を待つ間に、参加者たちはマイクロピペットの操作方法を練習した。
 ダイヤル式のプランジャーを回して容量を細かく調節する。
ダイヤル式のプランジャーを回して容量を細かく調節する。
マイクロピペットは、μℓ(マイクロリットル/100万分の1ℓ)単位の微量の液体を計量するのに適した器具で、先端に専用の使い捨てピペットチップを取り付けて使用する。
 ピペットチップは滅菌処理がされている。
ピペットチップは滅菌処理がされている。
一通り操作に慣れたところで、ドライアイスが溶け切ったことを確認し、用意されたバッファー(緩衝液)をマイクロピペットでサンプルに加える。

抽出用のバッファーは、界面活性剤とEDTA(エチレンジアミン四酢酸)を混ぜた溶液で、細胞膜を溶かしてDNAを核から取り出す作用がある。さらに、サンプル中に混入しているRNAを分解するため、RNase A(リボヌクレアーゼ)という酵素を加える。
試薬を混ぜたサンプルは、ボルテックスミキサーで撹拌する。
 ボルテックスミキサーのヘッドゴムにチューブの底を押し付けると、振動して内容物が混ざる。予想以上の強烈な振動が伝わってくる。
ボルテックスミキサーのヘッドゴムにチューブの底を押し付けると、振動して内容物が混ざる。予想以上の強烈な振動が伝わってくる。
撹拌したチューブは、細胞組織を溶かし、RNAを分解するため、ウォーターバスで10〜15分ほど温める。

加温後は、DNAからタンパク質を分離させる「カラム洗浄」と呼ばれる工程に移る。温めたチューブ内のサンプルは、若干色が変化しているようにも感じる。そこに酢酸や酢酸カリウム、塩化カリウムなどを混合したバッファーを加え、撹拌した後、氷の中に静置する。
 DNA抽出には温度変化を利用した工程が多い。
DNA抽出には温度変化を利用した工程が多い。
パーソナル・バイオラボ
「Bento Lab」を使ってみる
静置後、チューブ内のサンプルを二層式のフィルターチューブへ移し、遠心分離機にかける。使用するのは、DNA解析に必要な3つの機能を搭載した弁当箱サイズの装置「Bento Lab」だ。今回は”パーソナル・バイオテクノロジー”がテーマなので、DIYバイオのシーンの文脈で開発されたBento Lab を採用したという。
 遠心が始まると、モーター音が鳴り響き、参加者たちから感嘆の声が上がった。
遠心が始まると、モーター音が鳴り響き、参加者たちから感嘆の声が上がった。
チューブを中央に配置された遠心分離機にセットし、最大の8000xg(重力加速度の8000倍)で遠心する。
遠心分離後、チューブ下部に溜まった溶液(上清液)は、不純物が濾過されたため濁りが取れて透明に近づいている。フィルターを通り抜けて底に残ったペレット(不純物を含んだ沈殿物)を壊さないよう、この上清液だけをピペットで慎重に吸い出し、新しいチューブに移す。

次は、溶液内に溶けているDNAをまとめる工程だ。DNAを析出する作用を持つエタノール、タンパク質を変性させるグアニジン塩酸塩などが含まれたバッファーを加え、DNAを凝集させていく。加えるバッファーは、上清液の1.5倍量だ。
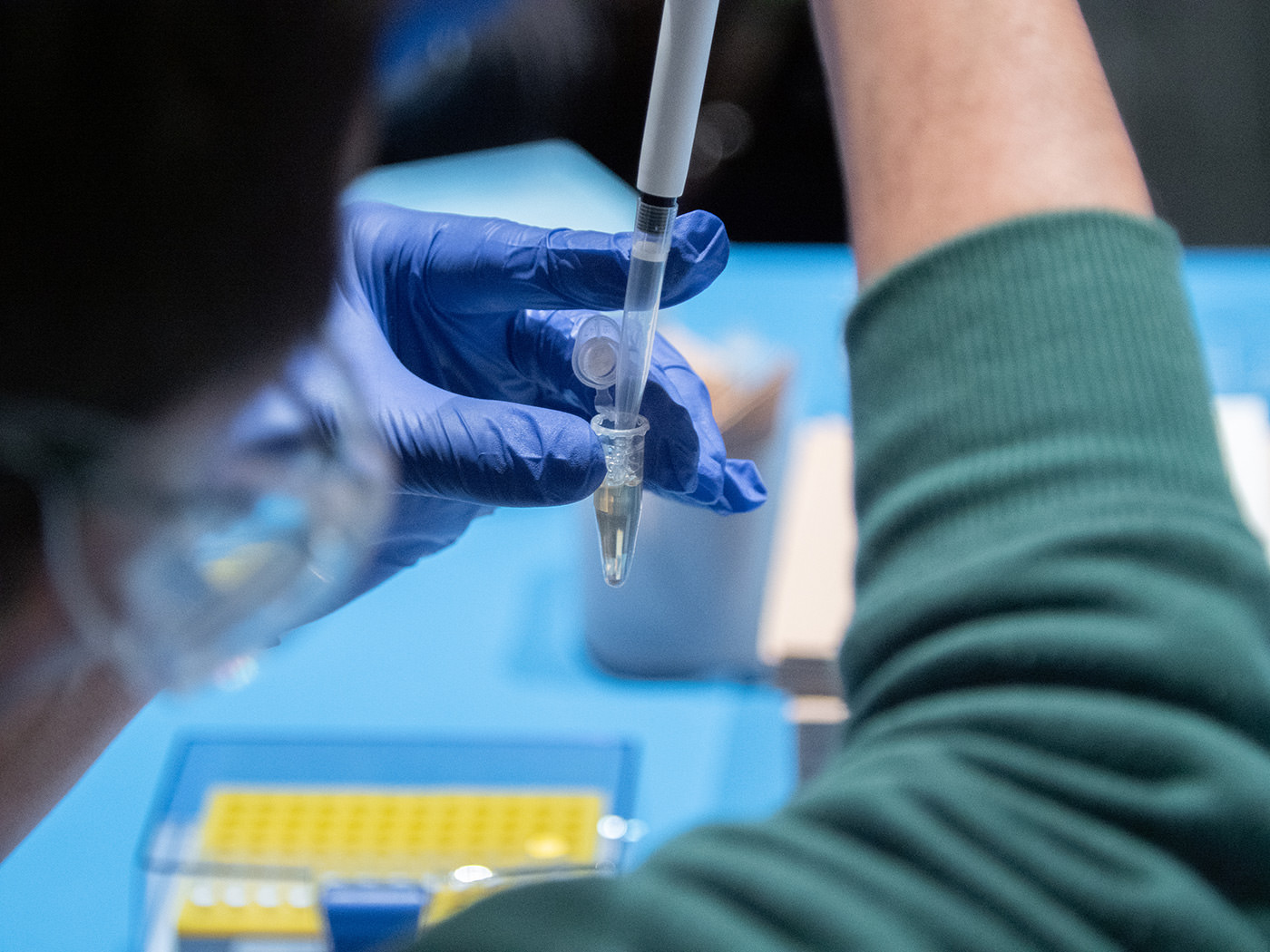 繊細に吸引・排出を繰り返す「ピペッティング」と呼ばれる操作でチューブ内を撹拌。よく混ぜ合わせる。
繊細に吸引・排出を繰り返す「ピペッティング」と呼ばれる操作でチューブ内を撹拌。よく混ぜ合わせる。
バッファーを混ぜた上清液400μℓを、ピペットで黄色のフィルターチューブに移す。これを再び Bento Lab で30秒遠心。今回は先ほどの工程とは逆に、DNAを捕捉しているフィルターの方を残し、濾過された液体は廃棄する。そこに再度、バッファーと上清の混合液を300〜400μℓ追加。同じ工程を繰り返して、DNAの収量を増やしていく。
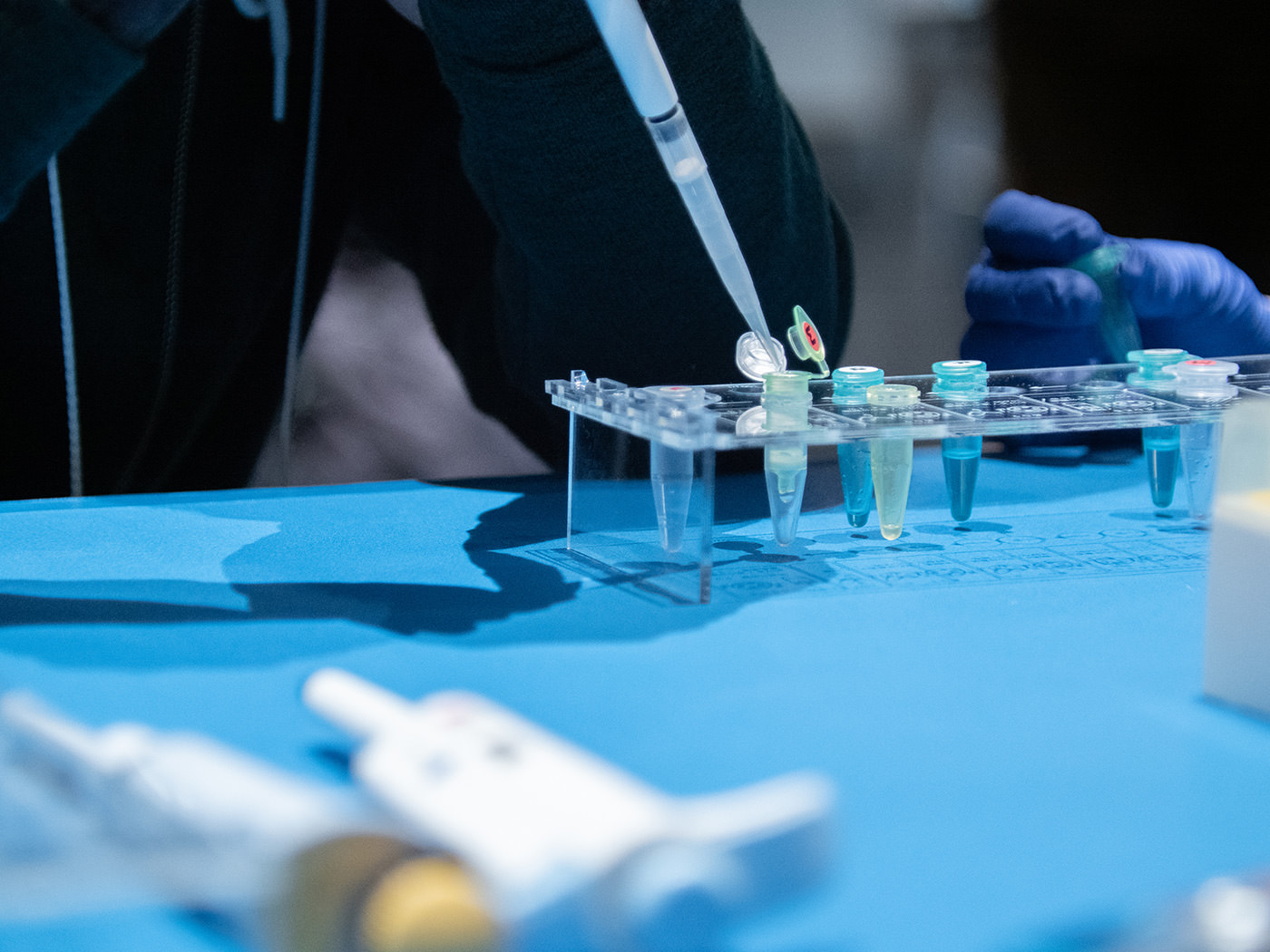 本来、この工程は1度で行うそうだが、Bento Lab のサイズの都合上、工程を一部変更し、2回に分けて行なった。
本来、この工程は1度で行うそうだが、Bento Lab のサイズの都合上、工程を一部変更し、2回に分けて行なった。 壇上から工程の変更について説明をする津田氏
壇上から工程の変更について説明をする津田氏
さらに、DNAを捕捉しているフィルターに対してエタノール洗浄と遠心分離を繰り返し、付着した不純物を取り除いていく。
 この頃になると、ピペットや機材の操作にも慣れ、参加者の間にはリラックスした空気が流れていた。
この頃になると、ピペットや機材の操作にも慣れ、参加者の間にはリラックスした空気が流れていた。
2度の洗浄の後、DNAが捕捉されたフィルターだけを取り出し、新しいマイクロチューブに移し替える。その後、DNAが水に溶ける性質を利用し、フィルターの上からDNAを溶出するバッファーを注入して、収量をあげるため5分間静置。DNA抽出の工程もいよいよラストに近づいてきた。
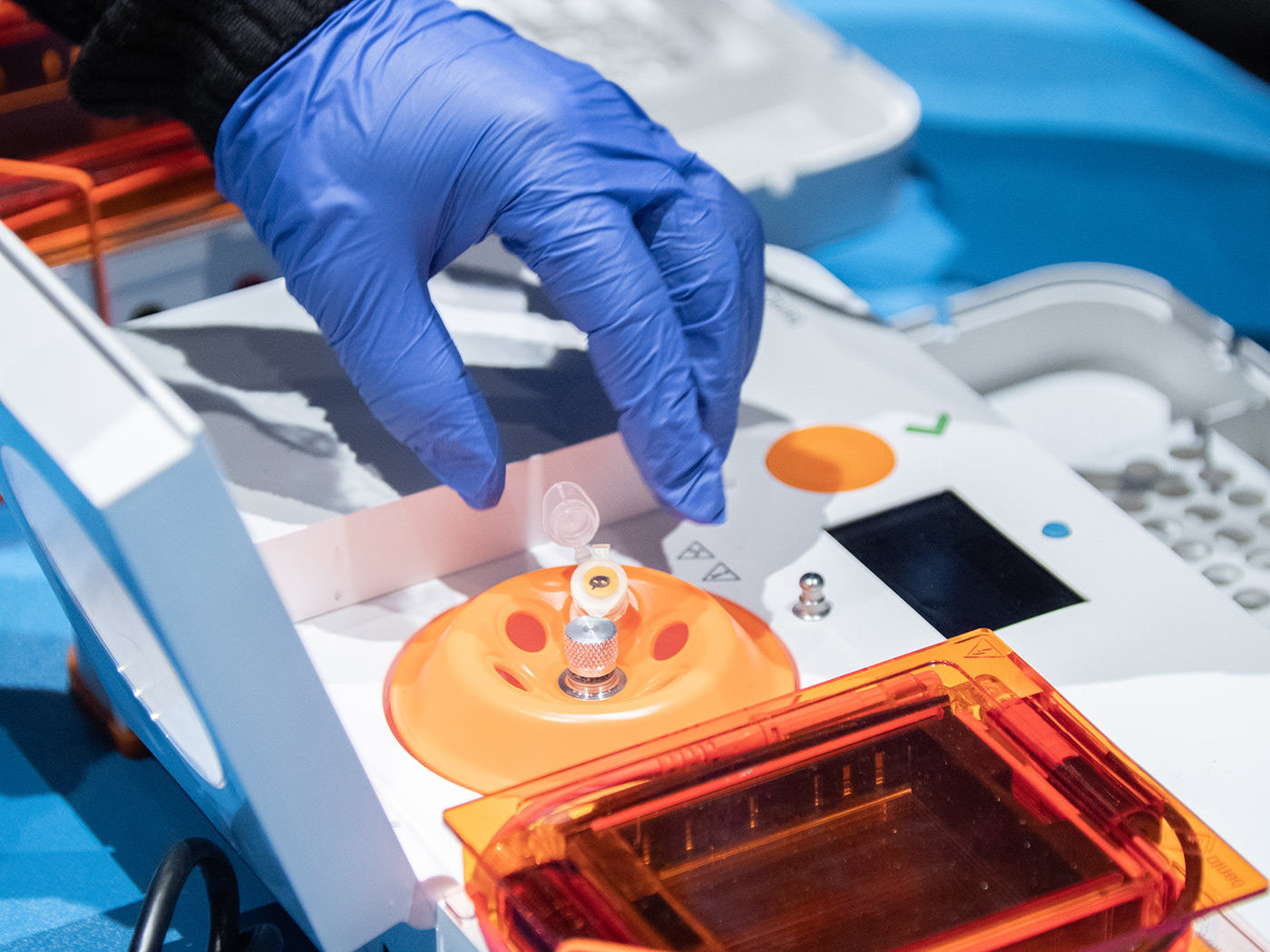
1分間、遠心分離。フィルターで濾過され、チューブの下部に溜まった透明の溶液を確認。この中にDNAが溶出しているという。

目視では確認できないので、あまり実感は湧かないものの、ここまででようやくDNA抽出の工程が終了。時刻はすでに16時を回っていたが、会場に安堵の空気が広がる。しかし、津田氏から「ここまでで全工程の35%ぐらいです」と伝えられると、達成感に包まれていた参加者たちもさすがに苦笑していた。
PCRで特定の領域の
DNAの量を増幅させる
続いて、DNAをPCRで増幅する工程に移る。PCR(polymerase chain reactionの略、ポリメラーゼ連鎖反応)とは、サンプルのDNAから特定の領域だけを増幅する方法のことだ。今回のPCRでは、葉緑体DNAの一部である「rbcL遺伝子」の領域を増幅させていく。
「DNAは、高温の環境にさらされると、二本鎖の結合が解けて一本鎖に分離し始めます(熱変性)。その後、少し温度を下げると、一本鎖になったDNAの特定部分にプライマーと呼ばれる20塩基ほどの短いDNA断片が結合します(アニーリング)。その後、再び温度を上げると、プライマーの結合部分を起点にDNAポリメラーゼという酵素がDNAの複製(伸長)を開始します。この3つの温度変化による反応を何サイクルも繰り返すことで、短時間でDNAを指数関数的に増幅させることができるのです」(津田氏)
プライマーは、増幅したいDNA領域にあわせて設計したフォワードプライマーとリバースプライマーの2種類を1セットとして使用する。PCRでは、このプライマーを増幅したいDNAと相補的になるよう設計することが重要だという。元になるDNA(鋳型DNA)の対象部分にプライマーが結合すれば、そこが目印となり、DNAポリメラーゼによってコピーがつくられていくからだ。
原理的には、このサイクルを1回終えるたびに、増幅したい領域のDNA量は2倍に増えていく。つまり、仮に30サイクル繰り返せば、元のDNA量をおよそ10億倍まで増やすことができるわけだ。
3D Animations - Experiments & Techniques: Polymerase Chain Reaction - CSHL DNA Learning Center
PCRでは、あらかじめ設定した通りに温度変化を起こすサーマルサイクラーという機材を使用する。今回は Bento Lab に付属する同機能を使ってPCRを行った。まずは、DNAを1本鎖にする温度(98℃)、アニーリングに適した温度(45-72°C)、DNAの伸長に必要な温度(72℃)のサイクルを30回繰り返すよう、サーマルサイクラーを設定。続いて、PCRに必要な反応液の作製に取り掛かる。
 10μℓ以下というごく微量の試薬を扱うため、一層慎重なピペット操作が必要だ。
10μℓ以下というごく微量の試薬を扱うため、一層慎重なピペット操作が必要だ。
反応液をつくるための試薬には、耐熱性のDNAポリメラーゼやDNA合成の材料となるdNTP(4種類の塩基に糖とリン酸が結合したもの)などを混合させた「PCRマスターミックス」を利用した。この試薬と、あらかじめ設計済みのプライマー、精製水を順番に加えていく。
 集中力を要する作業の連続だが、息のあったチームワークで工程を進めていく
集中力を要する作業の連続だが、息のあったチームワークで工程を進めていく
最後に、DNA抽出の工程で回収したDNA溶液(鋳型DNA)を加え、反応液を完成させる。あれだけ苦労して取り出したDNA溶液だが、PCRに使うのはたったの1μℓのみ。最先端科学の裏側では、このように地道な努力の積み重ねが日々行われているのだろう。ここまでのプロセスを通じて、身を以てそう感じた。
完成した反応液の入ったチューブを、各チームがBento Lab のサーマルサイクラーにセットし、ボタンを押すと、無事にPCRが開始した。
 あちこちのテーブルで歓声と拍手が沸き起こる。しかしこれは全行程の半分に過ぎず、まだまだ道のりは遠い。
あちこちのテーブルで歓声と拍手が沸き起こる。しかしこれは全行程の半分に過ぎず、まだまだ道のりは遠い。
4トーク“Genetics for Everyone: The journey towards accessible bioscience”
フィリップ・ボーイング氏
Bento Lab が目指す
「誰もがバイオに取り組める世界」
PCRの工程が完了するまでには、およそ90分ほどの時間を要する。反応の終了を待つ間、Bento Lab の開発者であるフィリップ・ボーイング氏の講演が行われた。
 英国を拠点に活動するボーイング氏は、バイオテクノロジーを基軸にさまざまな表現活動を行うアーティスティック・リサーチ・フレームワーク「BCL」のメンバーでもある。
英国を拠点に活動するボーイング氏は、バイオテクノロジーを基軸にさまざまな表現活動を行うアーティスティック・リサーチ・フレームワーク「BCL」のメンバーでもある。
彼はコンピューターサイエンスを学んでいた大学時代、遺伝子操作によって感光性を与えた大腸菌でバイオフィルムをつくる研究のことを知り、「今後は生物もプログラム可能な時代になる」と考えたという。その後、合成生物学の世界大会「iGEM(International Genetically Engineered Machine competition)」の活動に携わるようになった彼は、ユニークなアイデアを持つ多くのバイオハッカーたちとの出会いを重ねる。Bento Labの共同開発者となるベサン・ウルフェンデンもその一人だ。
iGEMでの活動を通じて「DIYバイオの潮流を感じ取った」というボーイング氏は、自らもDIYバイオに取り組み始める。しかし、バイオを扱う研究には、高額の機材の揃ったラボが必要だ。世界各地のバイオハッカースペースを視察したが、ラボの設立は容易ではない。研究者ではなくても、いつでもDIYバイオに取り組める環境をつくるにはどうしたらいいのか――。
「 Raspberry Pi や Arduino などの登場で、電子工学はより身近なものになりました。ならば、バイオの世界にも同じようなプラットフォームが必要だろうと考えたのです」

そんな思いから「誰もが自宅で生物工学に取り組めるバイオラボのアイデアが生まれた」とボーイング氏。2012年からスタートした Bento Lab の開発には、資金面や技術面でさまざまなハードルがあったが、2018年度末からようやく安定的に出荷可能な状態を迎えたという。
「大事なのは、バイオ実験ができるハードウェアをつくることではなく、この Bento Lab を使って何をするかだと思っています。私たちはこれまで、Bento Lab プロジェクトの一環としてたくさんのワークショップを行なってきました。今後は、すぐに実験が始められるスターターキットやビデオを使ったオンラインチュートリアルを充実させ、生物工学を趣味や個人の活動として根付かせていきたい」
“Genetics for Everyone: The journey towards accessible bioscience” フィリップ・ボーイング氏
5実験精製、シーケンシング
不純物を取り除き、
純度の高いDNAを精製する
19時近く、無事にPCRが終了したことを確認。次のステップとして、PCR反応産物からDNAを精製する工程に入る。より正確にDNAを解析するため、PCRの工程で加えたプライマーや酵素などの不純物(夾雑物)を取り除き、DNAの純度を高めるのが目的だ。
DNA精製には、マイナスの電荷を持つDNAの性質を利用して分離・解析を行うゲル電気泳動が用いられることもある。しかし今回は、DNA断片に吸着する特殊なビーズと磁力によって精製する方法を試すという。
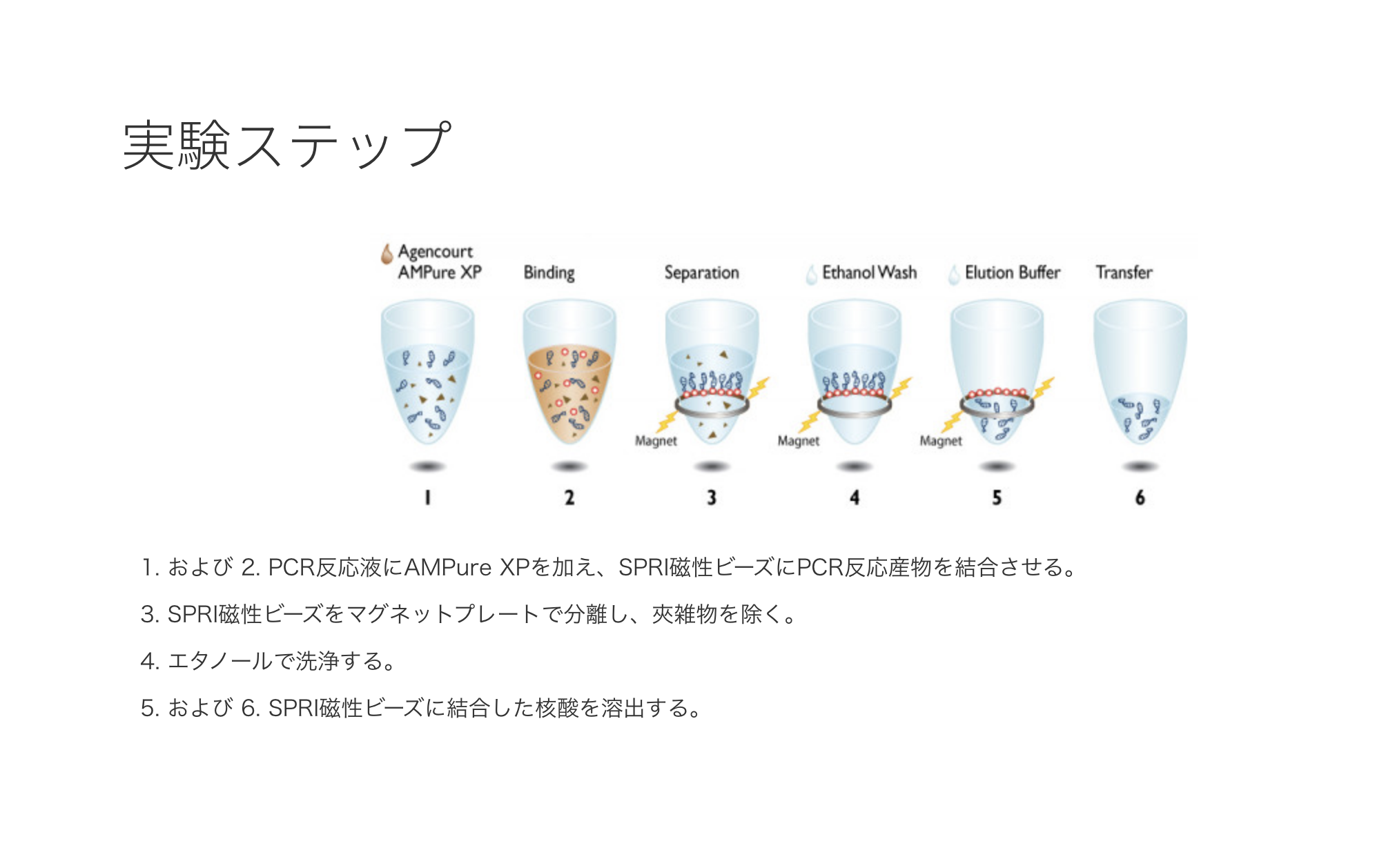 DNA精製試薬 AMPure XPの実験ステップ(ベックマン・コールターのウェブサイトより)
DNA精製試薬 AMPure XPの実験ステップ(ベックマン・コールターのウェブサイトより)
精製に使われる磁気ビーズは、表面を特殊なポリマーでコーティングされ、DNAを可逆的に結合することができる。また、結合するDNAのサイズが100塩基以上になるよう設計されているため、PCRの過程で使ったプライマーなど不要なDNA断片を除去できる利点もある。
まずは、サーマルサイクラーから取り出したPCRチューブに磁気ビーズの入った試薬(AMPureXP)を追加し、ピペッティングで混和。

今回のためにつくられたマグネットスタンドは、2本のチューブが立てられるようになっており、片側にのみマグネットが設置されている。この段階では、マグネットが付いていない方のスロットに挿して5分間静置。ビーズにDNAが結合するのを待つ。
 チューブ内の液体は、褐色透明に変化している。
チューブ内の液体は、褐色透明に変化している。
その後、チューブをマグネット側に移して2分間置くと、だんだんチューブ内の液体が透明になっていく。

よく見ると、チューブ内部のマグネットと接した部分に、焦げ茶色の小さな点状の物質が現れている。磁力によって磁気ビーズが壁面に吸い寄せられ集積したためだ。この固まりの中にDNAが含まれているはずだ。

夾雑物が残った液体をピペットで吸い出し、ビーズの固まりを壊さないようにエタノール洗浄を行う。2度繰り返すことで、不純物を確実に取り除いていく。

ここで、筆者のチームにはトラブルが発生。壁面に付いていたビーズの固まりが、チューブの底に落ちてしまったのだ。

そのため、洗浄に使ったエタノールを完全に吸い出せず、チューブ内に若干残ってしまった。これが後の工程に影響を与えないことを祈りつつ、作業を進める。
エタノールを除去したら、チューブをマグネットスタンドから外して精製水を加え、ピペッティングしながらしっかり混和。

再びマグネットスタンドに静置すると、磁気ビーズだけがマグネットに吸着し、DNAは水に溶け残った状態になる。つまり、溶液の中はほぼ水とDNAだけの状態だ。ビーズの固まりを避けて溶出液をピペットで回収し、新しいチューブに移動させれば、DNA精製の工程は完了だ。
精製したDNAの濃度や純度を測定する
精製したDNAは、分光光度計という機材でDNA量の計測(定量)を行う。つまり、精製したDNA溶液の中にどれぐらいDNAが含まれているかを調べるわけだ。
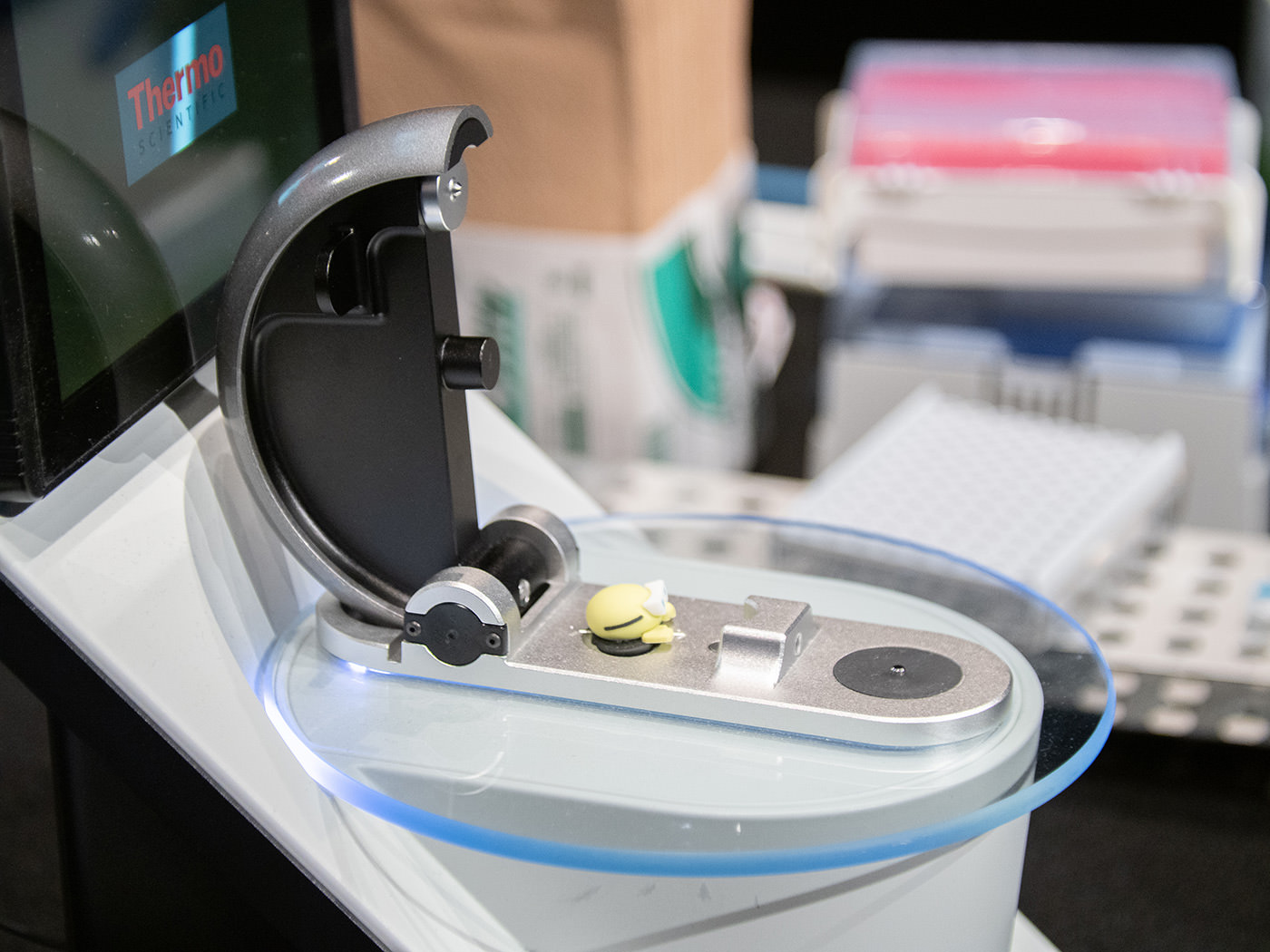 定量に使った「NanoDrop OneC 分光光度計」
定量に使った「NanoDrop OneC 分光光度計」
分光光度計では、ごく微量のサンプル溶液に特定の波長の光を当て、その透過率(吸光度)を調べることができる。DNAは、光の波長が260nm(ナノメートル)のときに最も良く光を吸収することがわかっており、この値からDNA濃度を算出することができるのだ。
一方、タンパク質は280nmの波長の光を最も良く吸収するため、もしタンパク質の混入があれば吸光度にも影響が出る。つまり、260nmの値に対する280nmの値の比を見れば、DNAの純度がわかる。目安として、この値が1.8以上あれば、高純度のDNAと判断できるという。
さらに、DNAの吸光度が最小になる波長は230nmとされているので、260nmの最大値と230nmの最小値を比較すれば不純物の混入(コンタミネーション)も調べられるそうだ。この比率が1を下回っていると、混入が起きている可能性が高いとされる。
DNA定量の工程では、この3つの値を調べ、シーケンシング(解析)が可能かどうかを判断していく。
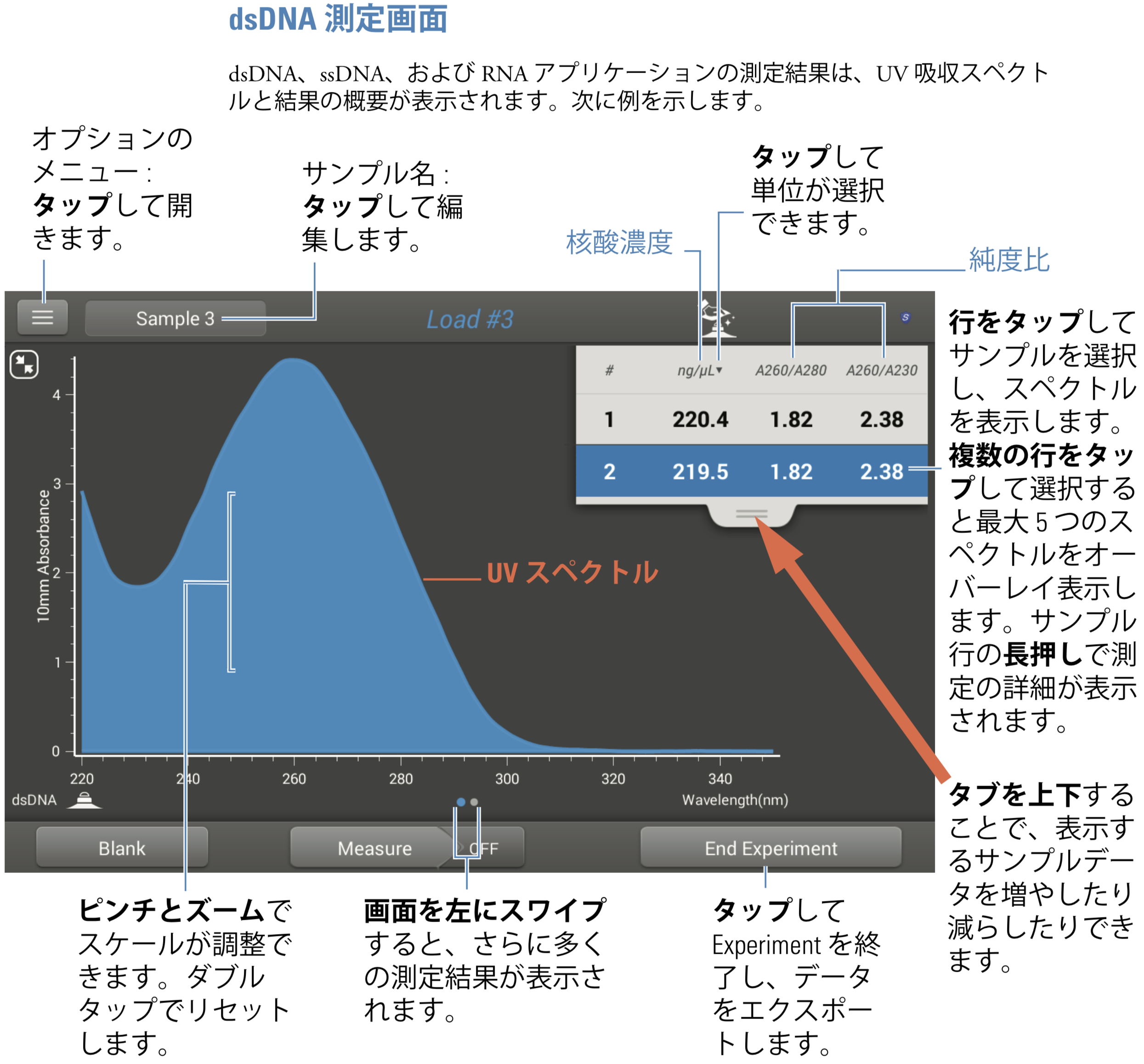 NanoDrop One のDNA測定画面(NanoDrop Oneユーザーガイド p.20 より)。結果はこのようなスペクトルで表示される。
NanoDrop One のDNA測定画面(NanoDrop Oneユーザーガイド p.20 より)。結果はこのようなスペクトルで表示される。
ここで津田氏より、部分的なプロトコル変更が伝えられる。全16チームのうち、食材を選んだ4組(白菜、にんじん、レンコン、ひよこ豆)については、葉緑体DNAの一部(rbcL遺伝子)を読むのではなく、ゲノムDNA全体の解読に挑戦することになったのだ。そのため4組のチームは、DNA抽出液(PCR試薬を追加する前の状態)を使って定量を行うことになった。
計測は各チームごとに行われた。1組ずつ壇上に設置された分光光度計のもとに集まり、DNA溶液(2μl)を台座に滴下する。アームを下げると、自動的に測定が始まる。
 まず精製水の値を基準値として分光光度計に登録。DNA溶液のそれぞれの値と比べることで濃度や純度を調べていく。
まず精製水の値を基準値として分光光度計に登録。DNA溶液のそれぞれの値と比べることで濃度や純度を調べていく。
各チームとも、まるで我が子の晴れ舞台を見守る親のような面持ちで測定結果を待つ。緊張の瞬間だ。結果は、わずか数秒程度で画面上に表示される。
 会場のスクリーンに映し出された結果を見て、一喜一憂する参加者たち。
会場のスクリーンに映し出された結果を見て、一喜一憂する参加者たち。
筆者のチームが選んだ「にんじんサラダ」は、芳しくない結果に終わった。DNAの濃度は、1μℓ当たり1.6ng(ナノグラム)。純度も基準値の1.8を下回った。気になるのは不純物の混入を示す値で、ボーダーラインとなる「1」を下回っている。やはり、エタノール除去が不完全だったことが影響していたかもしれない。率直に残念だった。
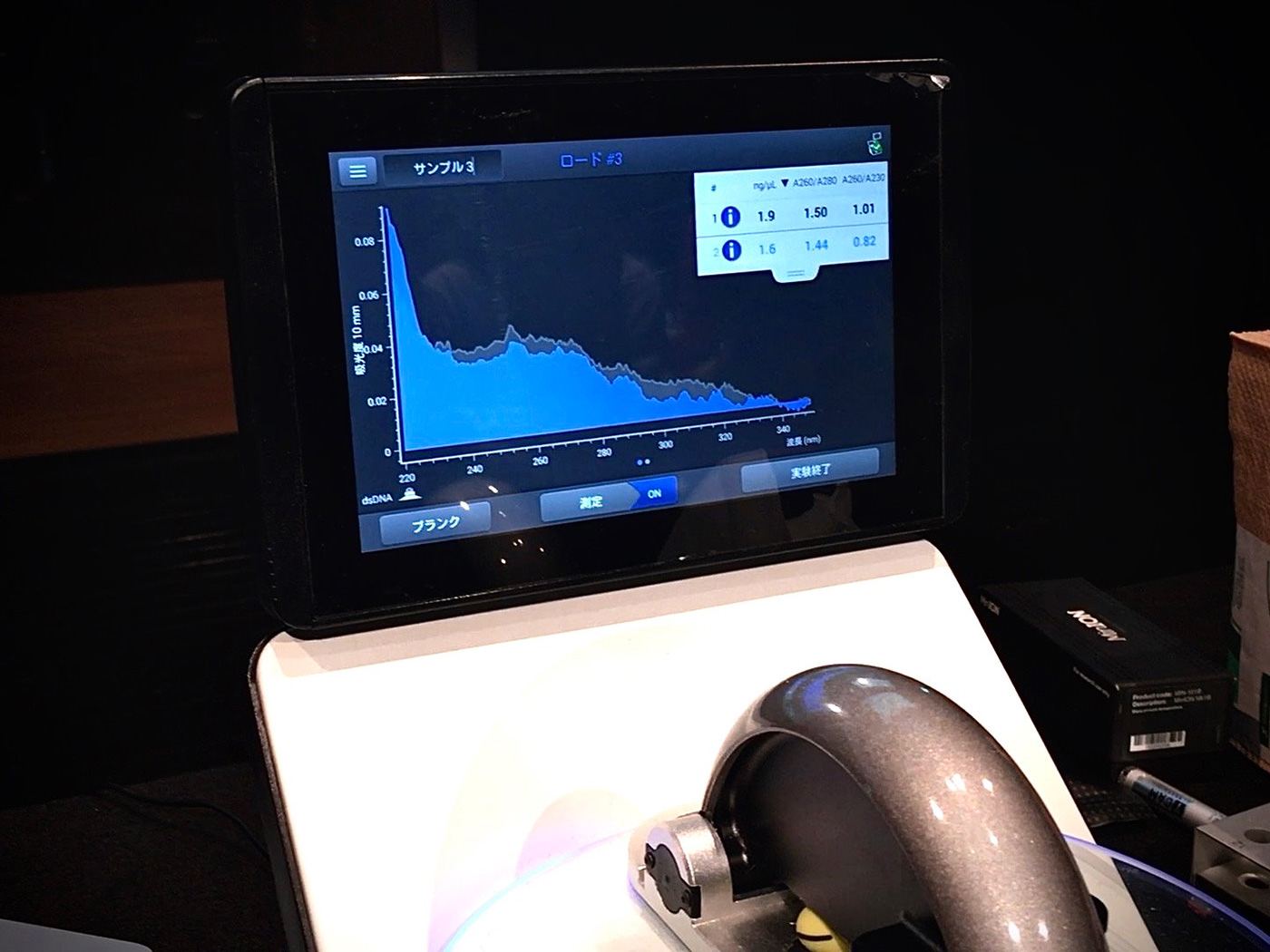
高い測定値が出るたびに「おお〜」というどよめきが上がり、会場はややコンペティティブな雰囲気に。結果が思わしくないチームは、落胆の表情を浮かべていた。

一方、計測を終えた参加者たちからは「どの工程で操作を間違ったのか」「素材によって抽出のしやすさに差があるのか」などの疑問が多く聞かれた。「計測結果から実験工程の適否を振り返る時間が欲しかった」と話す参加者も印象的だった。
DNAの定量開始からおよそ30分、16チームすべての結果が出揃った。チームによって、スコアに大きなばらつきがあるのは意外だった。そこで今回は、食材サンプルのカテゴリー(以下参照)ごとに、それぞれスコアが良かった上位2チームを選ぶことになった。
-
葉緑体DNAの一部を読むチーム(素材サンプル)……にんじん、トマト
-
葉緑体DNAの一部を読むチーム(料理サンプル)……炊き込みごはんの具、漬物
-
ゲノムDNA全体を読むチーム……白菜、ひよこ豆
津田氏から結果が発表されると、会場からは互いの健闘を讃えるかのように大きな拍手が湧き起こった。
最新式のシーケンサーでDNAの配列を読む
いよいよ本日最後となるDNAシーケンシングの工程に入る。今回は、選ばれた上位6チームのDNAサンプルをシーケンスするため、新たに6人1組のチームが組み直された。筆者のチームの「にんじんサラダ」は落選したため、「トマト」のチームにジョインして解析を行うことになった。時刻はすでに21時過ぎ。ここまで夕食も取らず、集中して作業してきた参加者たちの表情には、さすがに疲労が色濃く現れている。
各テーブルには1台ずつ、計6台のラップトップが置かれ、シーケンサーと接続されていた。今回使用する機材は、英国のオックスフォード・ナノポアテクノロジー社が開発した手のひらサイズのシーケンサー「MinION(ミナイオン)」だ。

シーケンサーで読み出された塩基配列は「リード」と呼ばれるが、従来のNGSと呼ばれるシーケンサーは、断片化したDNAを大量に読み取り、それらを大規模に並列に解読して「ショートリード」データを出力するものが主流だった。一方、MinION では、ナノサイズの穴(ナノポア)にDNAを一分子ずつ通し、その際に発生する電流の塩基ごとの微細な違いを利用して塩基配列を解読する。1本のDNAを断片化せず、頭から順番に解読していくため、短時間で長い配列を読む「ロングリード」データの出力が可能なのだ。
ちなみに、MinION の最大リード長は数十万bp(base pair:塩基対)に及ぶとされている。
MinIONを使うには、まず本体にフローセルをセットする。フローセルには、それぞれ4つの穴を持つ512チャンネルのナノポアが埋め込まれたセンサー付きの膜(センサーアレイ)が設置されている。その状態で本体をUSBでコンピュータに接続すると、専用のソフトウェア(MinKNOW)でフローセルの状態の確認ができる。
 フローセルは消耗品で、価格は10万円強だ。取り扱いには慎重さが求められる。
フローセルは消耗品で、価格は10万円強だ。取り扱いには慎重さが求められる。
フローセルの保管状態によってはは、使用できるナノポアが少なくなっている可能性がある。そのため、解析の前には必ずフローセルのチェックを行い、アクティブなナノポアの数を確認する必要がある。
続いて、PCRチューブの中でDNAサンプル溶液と試薬を混ぜ、DNAライブラリを調整する。DNAの末端(3’末端)にタグをつけ、そこにモータータンパク質を搭載したアダプターを追加するためだ。アダプターがナノポアに結合すると、モータータンパク質の作用でDNAの2本鎖は1本鎖に分離する。つまり、ナノポアにDNA分子を通すためには、このアダプターが重要な役割を果たすわけだ。

DNAにアダプターを結合させるため、サーマルサイクラーでインキュベートする。その後、ローディングビーズやバッファーも追加する。

疲労と空腹の中、工程の終わりが見えてきた興奮からか、会場は高揚感とハイテンションに包まれ、工程が進むたびにあちこちで歓声や拍手が沸き起こる状況に。インキュベート終了の際には、周囲で見守る参加者たちからカウントダウン・コールが行われ、お祭りのような雰囲気を醸し出していた。
一方、DNAライブラリとは別に、フローセルの準備も進める。プライミングポートを開け、ピペットの先を挿入したら、繊細なピペット操作で内部の空気を取り除く。その後、気泡が入らないように注意しながら、慎重に充填液(プライミングミックス)を注入していく。

次に、SpotONサンプルポートのカバーをゆっくりと外し、注入口を露出させる。ただ、この時点ではまだDNAライブラリは投入せず、プライミングポートから再びバッファーを少量追加する。このとき、バッファーをサンプルポート側から溢れさせないよう注意する。

そして最後にして最大の難関、DNAライブラリを注入する工程に移る。ここで失敗すると10万円のフローセルが無駄になってしまうリスクがあるため、注入を担当する参加者にはプレッシャーがのしかかる。
PCRチューブの中でDNAライブラリを何度かピペッティングしてから、肘をしっかり固定し、SpotONサンプルポートにゆっくり滴下していく。ポートから液体が溢れないよう、一滴ずつ送り込むイメージだ。テーブルを囲む参加者も全員、固唾を飲んで手元を見守る。

全量が入ったら、SpotONサンプルポートのカバーをしっかりと閉め、プライミングポート、MinION本体カバーの順に閉じていく。コンピュータ上でMinKNOW がデバイスを認識したら、START RUN を実行。数分後にはシーケンシングが始まった。
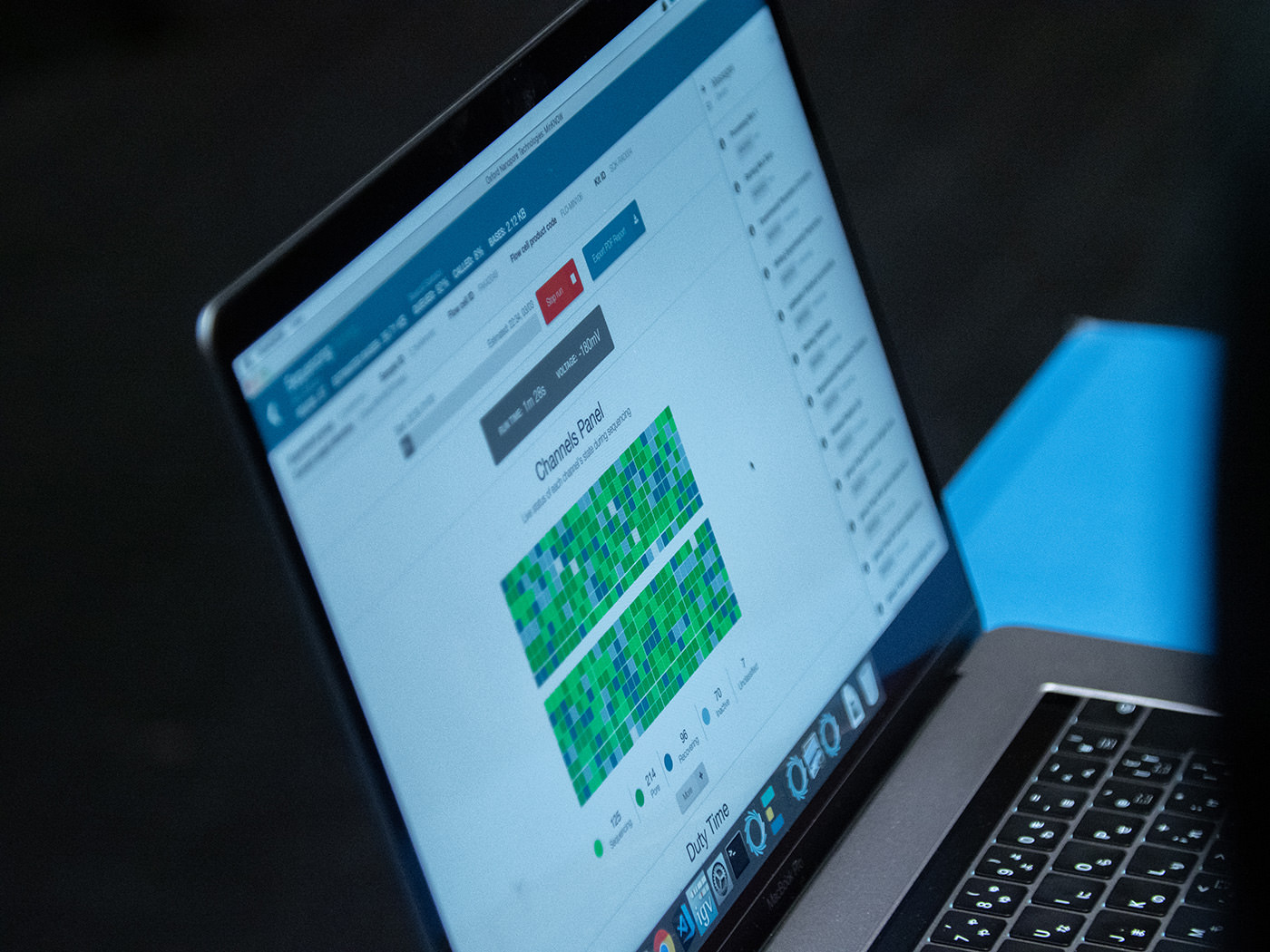
ファシリテーターの菅沼氏から、全行程が終了したことが伝えられると、会場には感慨と達成感が入り混じった大きな拍手が巻き起こった。ここに至るまでに要した時間は、ほぼ10時間。なかなかハードな工程ではあったが、バイオの知見を持たない初心者が、わずか10時間でDNA抽出からシーケンシングまでこぎつけたことは、瞠目すべき成果と言えるだろう。シーケンシング はこのまま翌朝まで続けられ、詳しい解析は翌日の午前中に行われる予定だ。
ワークショップ終了後も、コンピュータの画面には青や緑の光が明滅し続けていた。これはある意味、目に見えないDNAが可視化された姿といえる。小さなチューブの中の液体を覗いても実感できなかったその存在が、急に目の前に現れた気がして感慨深い。あらゆる生物の命を支える見えない生命システムを体感した、貴重な1日となった。
続きを読む
DAY2
「書き」・「生命倫理」“Write” and “Bioethics”
DNA合成やゲノム編集といったDNAの情報を書く技術、さらにそれらの技術に伴う生命倫理の問題について、レクチャーやワークショップから考えます。
レポートを読む































































