2019年3月1日〜3日、バイオテクノロジーの基礎と応用可能性を学ぶ3日間の集中ワークショップ「YCAM InterLab Camp vol.3」が開催された。本記事では、このイベントの2日目の模様をお届けする。1日目の記事からご覧になりたい方はこちら、イベントの概要を知りたい方はトップページをご覧いただきたい。
contact Gonzo
2019年3月1日〜3日、バイオテクノロジーの基礎と応用可能性を学ぶ3日間の集中ワークショップ「YCAM InterLab Camp vol.3」が開催された。本記事では、このイベントの2日目の模様をお届けする。1日目の記事からご覧になりたい方はこちら、イベントの概要を知りたい方はトップページをご覧いただきたい。
ワークショップ2日目の開始時刻は午前9時半。午前中は、昨夜一晩かけてシーケンシング(DNAを構成する塩基配列を読む作業)したDNAデータを解析する作業がメインとなる。
この日のプログラムも contact Gonzo によるエクササイズからスタートした。彼らから出されたお題は「参加者全員でランダムにぶつかり合う」というもの。ぶつかる時は声を出さず、できるだけ無表情で行わねばならないという。つまり、ぶつかられた部位や衝撃の強さだけで互いにコミュニケーションを図る試みと言える。
 会場を歩きながら、腕や肩、背中など体のさまざまな部位を使って互いに接触する。
会場を歩きながら、腕や肩、背中など体のさまざまな部位を使って互いに接触する。
接触を繰り返すうちに妙な信頼感が芽生え、次第にぶつかり方が激しくなっていく。ここが満員電車なら間違いなくトラブルになる当たりの強さでも、なぜか相手に負の感情は湧いてこない。脳が「攻撃」や「暴力」と認識していないからだろうか。悪意のないぶつかり合いを重ねることでパーソナルスペースが融解し、相手との心理的な距離感が縮まるメカニズムは非常に興味深い。
 参加者に指示を出す「contact Gonzo」の塚原氏
参加者に指示を出す「contact Gonzo」の塚原氏
続いて、塚原氏は参加者を3つのグループに分け、目を閉じた状態で一列になるように指示を出す。今度は互いに接触してはならず、それぞれ声を発したり手を叩いたりしながら、音の情報だけを頼りに列をつくっていく。言うなれば、イルカやクジラが音の反響によって周囲の状況を認識する「エコーロケーション(反響定位)」の擬似体験のようだ。
 積極的に動き回る人やその場を動かない人など、動き方にも個性が出る。
積極的に動き回る人やその場を動かない人など、動き方にも個性が出る。
音だけを頼りに移動するのは非常に難しく、どのグループもきれいな列はつくれなかった。しかし、視覚や触覚をシャットアウトすることで、ふだん使っていない脳の領域が賦活される感覚は、新鮮で心地よい。contact Gonzo の「知覚をバグらせる」エクササイズに、前夜の疲れがリセットされた気がした。
 最後は全員で一本の列をつくるワークにも挑戦。最終的に、団子状の集団から3本の列が伸びる不思議な形が出来上がった。
最後は全員で一本の列をつくるワークにも挑戦。最終的に、団子状の集団から3本の列が伸びる不思議な形が出来上がった。10時過ぎ、いよいよゲノムデータの解析に取り掛かる。壇上には、ライフサイエンス統合データベースセンター(DBCLS)特任助教を務める片山俊明氏と、京都大学大学院医学研究科 ビッグデータ医科学分野准教授の鎌田真由美氏が登場。ゲノムの「読み」において重要な役割を果たす「バイオインフォマティクス」について説明が行われた。
 ゲノム情報を解析するソフトウェア開発のほか、国際会議 BioHackathon(バイオハッカソン)も主催する片山俊明氏(左)と、ゲノム医科学や医療ビッグデータ解析を専門とする鎌田真由美氏(右)
ゲノム情報を解析するソフトウェア開発のほか、国際会議 BioHackathon(バイオハッカソン)も主催する片山俊明氏(左)と、ゲノム医科学や医療ビッグデータ解析を専門とする鎌田真由美氏(右)
バイオインフォマティクスとは、バイオ(生物学)とインフォマティクス(情報学)が融合した新しい分野で、膨大な生命関連データをコンピュータによって整理し、解析する学問領域だ。片山氏はこの分野の専門家として、これまで生命情報科学用ライブラリ『BioRuby』の開発や、ヨコヅナクマムシのゲノム解析プロジェクトなどに携わってきたという。
バイオインフォマティクスが登場した背景には「シーケンサーの著しい進化がある」と片山氏。第一世代のシーケンサー(キャピラリー型)は1990年代に登場し、国際ヒトゲノム計画でも活躍したが、電気泳動によって一つ一つ配列を決めていくため、膨大な時間とコストがかかる点が課題だったという。
「しかし、2000年代半ば以降、塩基配列を高速に解読できる次世代シーケンサー(NGS)が次々に登場します。その結果、さまざまな生物のゲノム解読が進み、得られる生物データ量も飛躍的に増加しました」(片山氏)
つまり、それらのデータを効率的に処理し、解析するための手段として開発されたのがバイオインフォマティクスというわけだ。
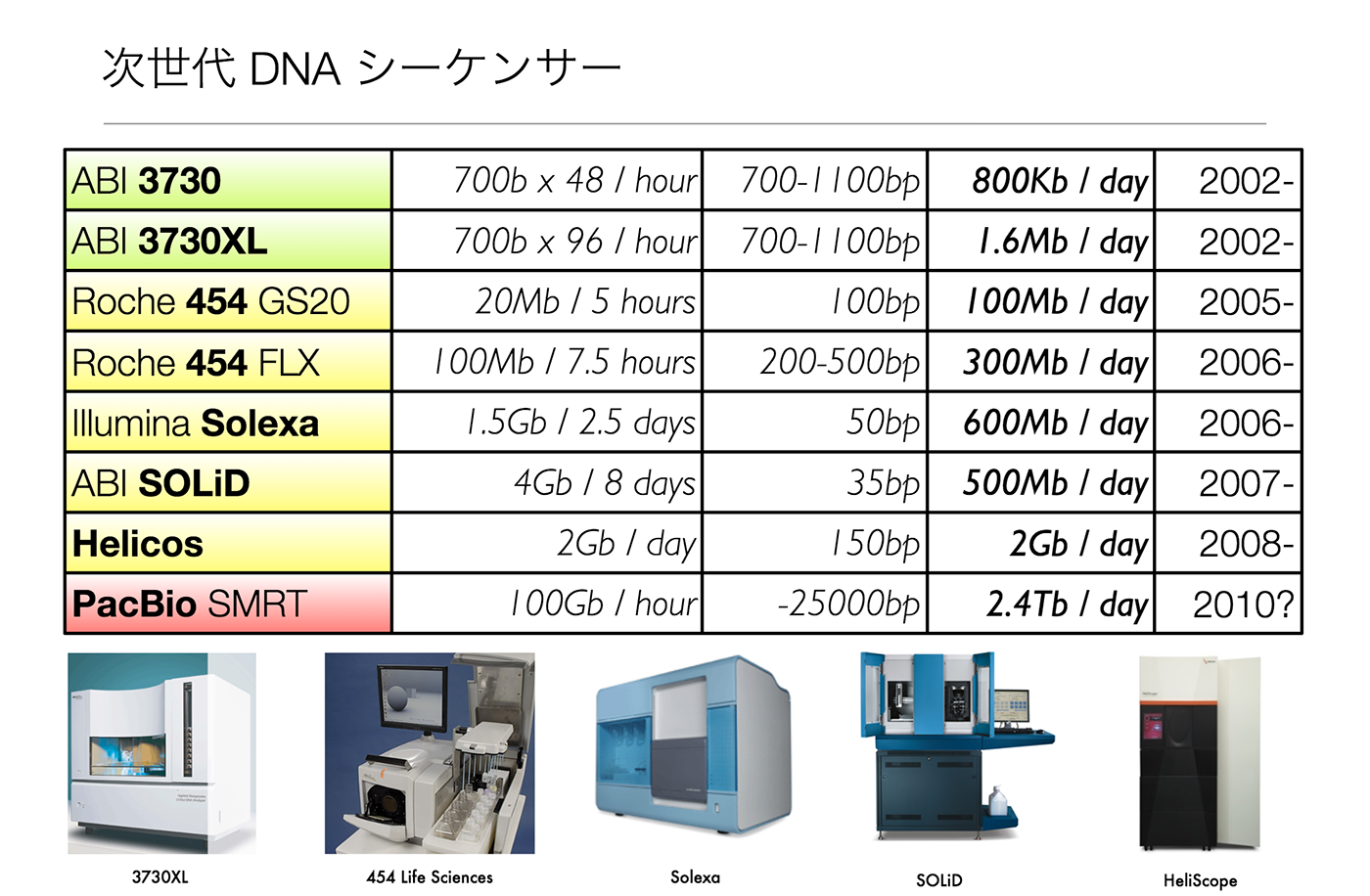 次世代シーケンサーの処理能力は、現在も劇的な進化を遂げている。米国Illumina社やスイスRoche社が二大メーカーとして知られる。(片山氏のプレゼン資料より)
次世代シーケンサーの処理能力は、現在も劇的な進化を遂げている。米国Illumina社やスイスRoche社が二大メーカーとして知られる。(片山氏のプレゼン資料より)
「ゲノム配列を決定するには、シーケンサーで読みだした配列の断片を大量に集め、それらをジグソーパズルのようにつなげながら元の形に戻す作業(アセンブリ)が必要です。一方、塩基配列のデータそのものは、ATGCのランダムな羅列です。そのため、どこにどんな遺伝子があるのか、近縁の生物種と配列に共通性はあるのか、遺伝子から産出されるタンパク質にはどのような機能があるかなどを多面的に調べる必要があります」(片山氏)
このとき参照されるのが、データベースに登録されているアノテーションと呼ばれる情報だ。これはゲノムに付随する重要な情報のことで、新しく解明されたアノテーション情報は随時データベースに登録され、リファレンス情報として重要な役割を果たすという。
バイオインフォマティクスの技術は、こうしたデータベースの構築にも欠かせないものだ。当初はDNAの塩基配列決定や遺伝子の探索などの目的に用いられてきたが、現在は解読されたゲノム情報をもとに、遺伝子から転写されるRNAの解析(トランスクリプトーム解析)や、翻訳産物であるタンパク質の配列や機能の解析(プロテオーム解析)などへの活用が進んでいる。
実際、ヨコヅナクマムシのゲノム解析プロジェクトでは、ゲノム配列の決定や遺伝子の同定だけでなく、クマムシの極限環境における耐性を調べるためトランスクリプトーム解析まで行なったそうだ。
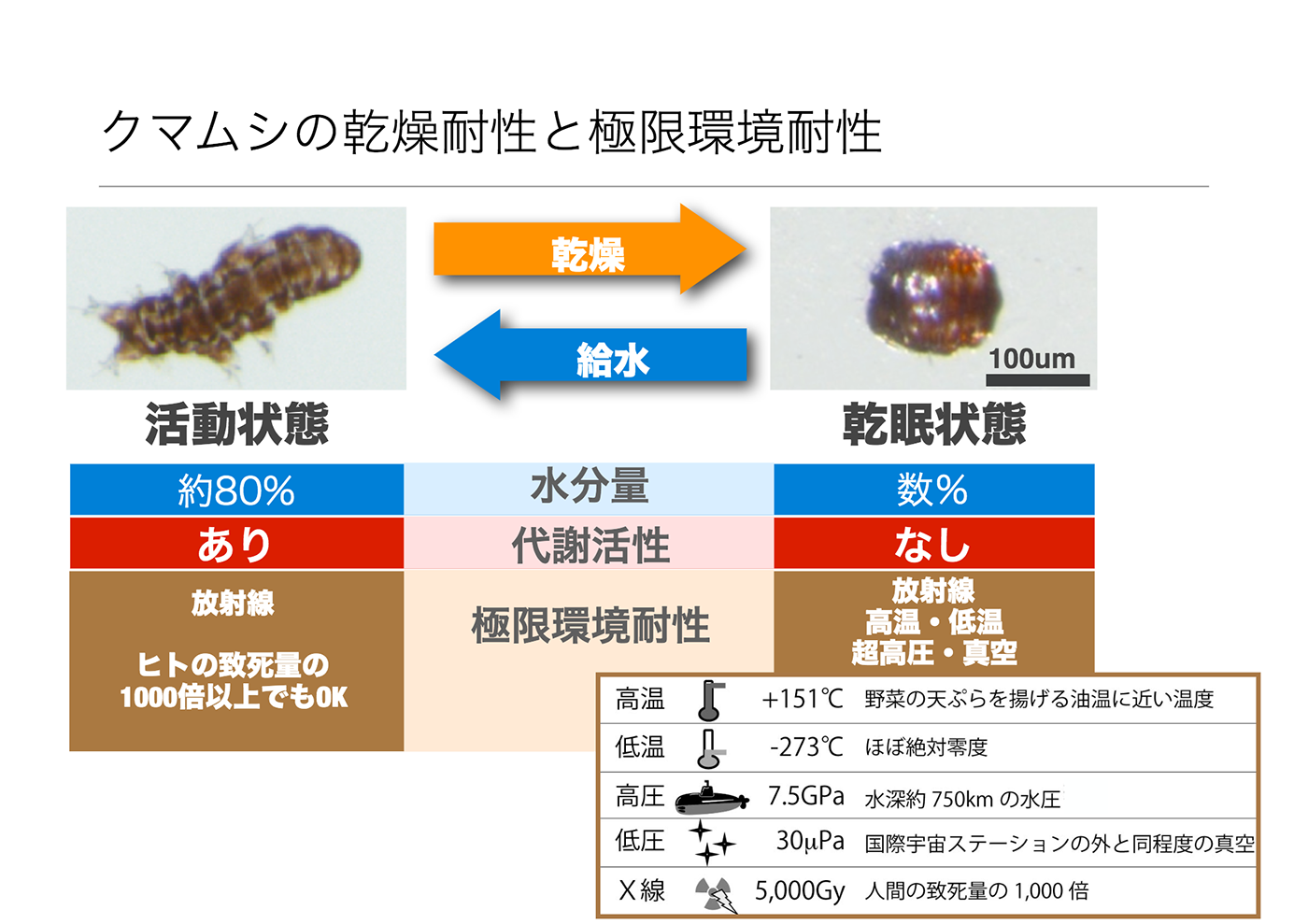 クマムシは、地球上のあらゆる場所に生息する体長0.5mm~1mm程度の生物。極度の乾燥状態では「乾眠」と呼ばれる仮死状態となり、ほぼ絶対零度の超低温や真空状態、放射線などに強い耐性を発揮することで知られている。(片山氏のプレゼン資料より)
クマムシは、地球上のあらゆる場所に生息する体長0.5mm~1mm程度の生物。極度の乾燥状態では「乾眠」と呼ばれる仮死状態となり、ほぼ絶対零度の超低温や真空状態、放射線などに強い耐性を発揮することで知られている。(片山氏のプレゼン資料より)
一方、バイオインフォマティクスを活用した取り組みの中で、とくに大きな注目を集めているのは医科学への応用だ。
「ゲノム情報は、さまざまな遺伝的な疾患に関与しています。まず、全ゲノムがわかれば、染色体異常や遺伝子変異などゲノムの異常に由来する疾患の発症を知ることができます。また、正常な細胞とがん化した細胞の遺伝子の発現(タンパク質をつくること)状況を比べて、細胞のがん化のメカニズムを調べることもできる。さらに、ゲノム情報と疾患・臨床情報を紐づけてデータベース化することで、より個人にあわせた医療(個別化医療)につなげていく取り組みも始まっています」(片山氏)
 ライフサイエンス統合データベースセンター(DBCLS)特任助教・片山俊明氏
ライフサイエンス統合データベースセンター(DBCLS)特任助教・片山俊明氏
ヒトゲノム計画完了後、ゲノム配列には個人ごとに違い(一塩基多型)があり、体質や病気へのかかりやすさなどに影響を与えることがわかってきた。欧米では、ゲノム情報と疾患・臨床情報を紐づけたデータベースが急速に整備されつつあり、すでに医療現場での診断や治療に役立てられているそうだ。
「だからこそ、日本のゲノム医療の進展には、情報基盤となる日本人のゲノム情報に特化したデータベースの構築が重要」と強調する片山氏。そんな彼自身も、2018年に公開された日本人の全ゲノム変異を統合したデータベース『TogoVar』の開発に携わっている。
また片山氏は、この日のワークショップのサポーターとして、東北大学 東北メディカル・メガバンク機構の荻島創一教授を紹介。荻島氏は、日本人およそ3500人分の全ゲノム情報とコホート調査による健康調査情報を統合したデータベース『dbTMM』の開発プロジェクトを主導した一人だ。つまり今回のワークショップでは、日本のゲノム医科学をリードする研究者たちから直々にサポートを受けられるわけだ。なんとも贅沢な企画である。
 各テーブルを回り、参加者たちの解析のサポートに奔走する東北メディカル・メガバンク機構の荻島創一教授(右)
各テーブルを回り、参加者たちの解析のサポートに奔走する東北メディカル・メガバンク機構の荻島創一教授(右)
今回、解読の対象となるのは「ゲノム弁当」に使われた食材だ(DAY1のレポート参照)。「ゲノム弁当」は、2016年にYCAMで開催されたイベントの関連で、片山氏率いるバイオハッカソン・チームとYCAMメンバーの共同企画ではじめて実現した。
もともとゲノム解読済み食材で弁当をつくるアイデアは、2005年頃からゲノム関連の研究者たちの間で広がり始めたという。しかし当時、ゲノムが解読された食材はごくわずか。その後、2010年代以降になって野菜や穀物、肉類などのゲノム解読が一気に進展。具材が充実したことで、ようやく実現化したという。つまりゲノム弁当とは、シーケンサーの進化の賜物といえるプロジェクトなのだ。
DAY2のワークショップでは、MinION シーケンサーで読み出した ゲノム配列がどの生物に由来するものなのか、データベースを参照しながら検索する実習を行う。ゲノム解析のフローを整理してみると、以下のようになる。
DAY1のレポートでも触れたとおり、今回ゲノムを「読む」のは、以下の6つのサンプルだ。
① 全ゲノム配列が高い精度で完全決定済み(完全ゲノム)……白菜、ひよこ豆
② 全ゲノム配列がある程度の精度で決定済み(ドラフトゲノム)……にんじん、トマト
③ 複数の食材が混在する料理サンプル……炊き込みごはんの具、漬物
PCRを行う前のDNAサンプルを使ってゲノムを丸ごとシーケンスした①については、完全ゲノムデータ(リファレンス配列)にマッピング(アラインメント)し、同じ配列が得られているかどうか解析していく。
PCRでrbcL遺伝子領域を増幅したDNAサンプルを読んだ②と③については、DNAバーコーディング(ある特定の遺伝子領域の配列を解析することで生物種を同定する方法、DAY1レポート参照)を使って植物のメタバーコーディング解析を行う。
さっそく各チームは、パソコンでシーケンスの結果を確認した。シーケンサーで読み取られた塩基配列は「リード」と呼ばれるが、このリードがどのぐらい読み取れたかを MinION に付属するソフト「MinKNOW」のビューワでチェックする。
 トマトチームの結果。ロングリード がほとんど取れていない……?
トマトチームの結果。ロングリード がほとんど取れていない……?
図の横軸はリード長、縦軸はある範囲のリード長の総塩基数、つまりどのくらいの長さのリードがどのくらい読めているかという分布を表している。筆者の参加する「トマト」を読んだチームは、PCRで断片化したDNAサンプルをシーケンスしたため、短いリードのみが取れていたようだ。結果は、読み出したリード数が 922.36 K(約92.2万リード)、解読した総塩基数(リード長×リード数)は 390.02 Mbp(約390万塩基対)だった。
一方、シーケンスした6つのサンプルのリードデータは、すでにデスクトップ上のフォルダに格納されていた。MinION のリードデータは「FAST5」と呼ばれる特殊なファイル形式で出力される。同時に、MinIONでは付属ソフトが自動で「FASTQ」というテキスト形式のデータに変換してくれる仕様になっている。
今回解析に使うFASTQファイルとは、シーケンスデータの解析時に用いられる代表的なファイル形式の一つだ。FASTQファイルのリードデータは、1リードごとに 4 行で記述される。@で始まる1行目には配列ID、2行目に読み出された塩基配列、 3行目にはID または改行が記され、4行目にクオリティスコアが表示されている。
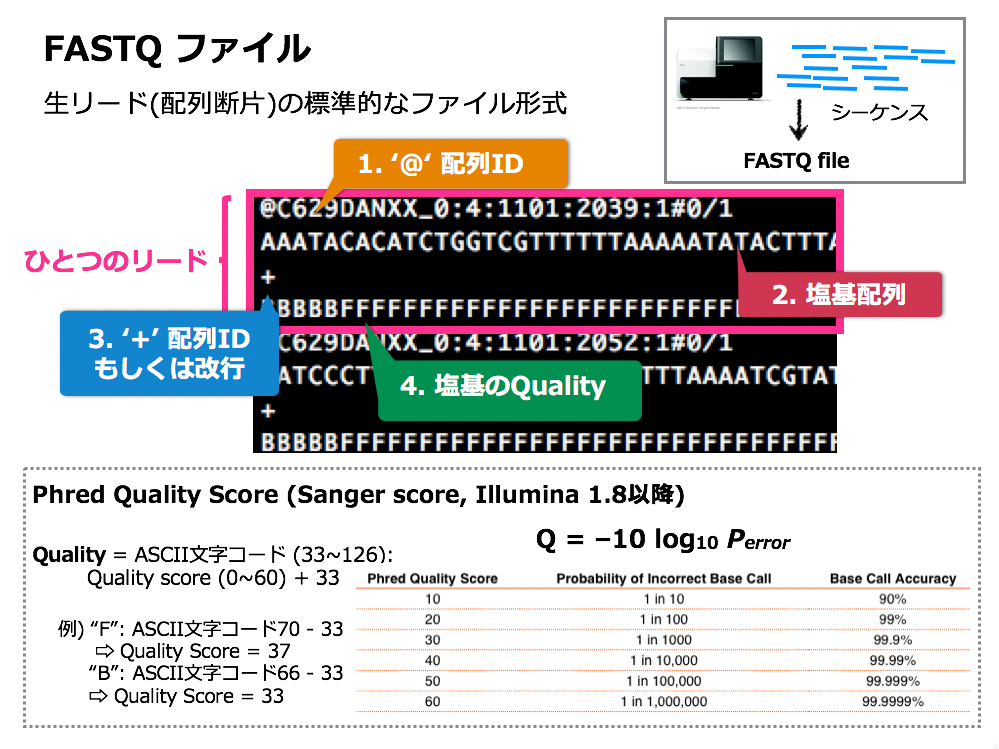 FASTQファイルのしくみ(片山氏のプレゼン資料より)
FASTQファイルのしくみ(片山氏のプレゼン資料より)
クオリティスコアとは、読み出された配列の信頼度を表す数値で、1塩基ごとに割り当てられている。わざわざ信頼度がセットになって出力されるのは、シーケンスの際に一定の確率でエラーが起きるためだ。
クオリティスコアは、エラーの発生率p を変換式(上図参照)に導入して得られた値Q として算出される。例えば、シーケンサーが100回に1回ほど間違った配列決定をしてしまうと仮定すると、そのエラー率(p)は 1%(p=0.01)だ。この場合、クオリティスコア(Q)は -10 × log10 (0.01) = 20 となる。つまり、クオリティスコアが 20 ならば、読み出された塩基配列の信頼度は 99% ということができる。
さらにクオリティスコアは文字列ではなく、アスキーコードで記述される。アスキーコードとは、整数(0~127)を英数字や記号などで表現する7ビットの文字コードのことだ。1塩基に1文字を割り当てる都合上、このようなスタイルが取られているようだ。
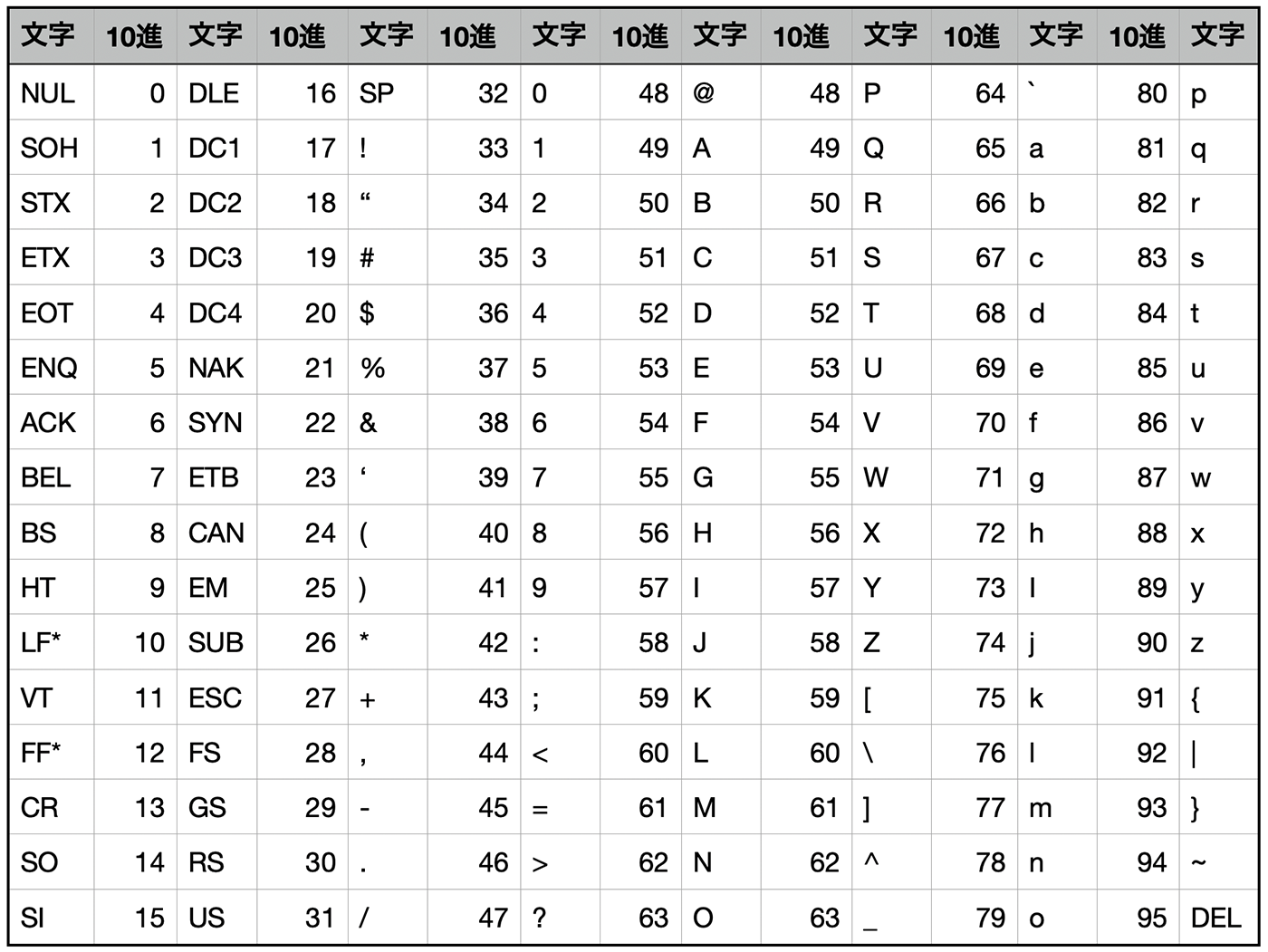 アスキーコードと英数字・記号はこのように対応している。
アスキーコードと英数字・記号はこのように対応している。
実際に記述されるアスキーコードは、クオリティスコアQ に 33 を足し合わせた数値に対応している。つまり、FASTQファイルのリードデータ上でクオリティスコアが「F」と表記されていた場合、F に対応する10進数は 70 であるため、クオリティスコアQ は 70 - 33 = 37 となる。
我々「トマト」チームも、さっそく読み出されたFASTQファイルを開いてみた。
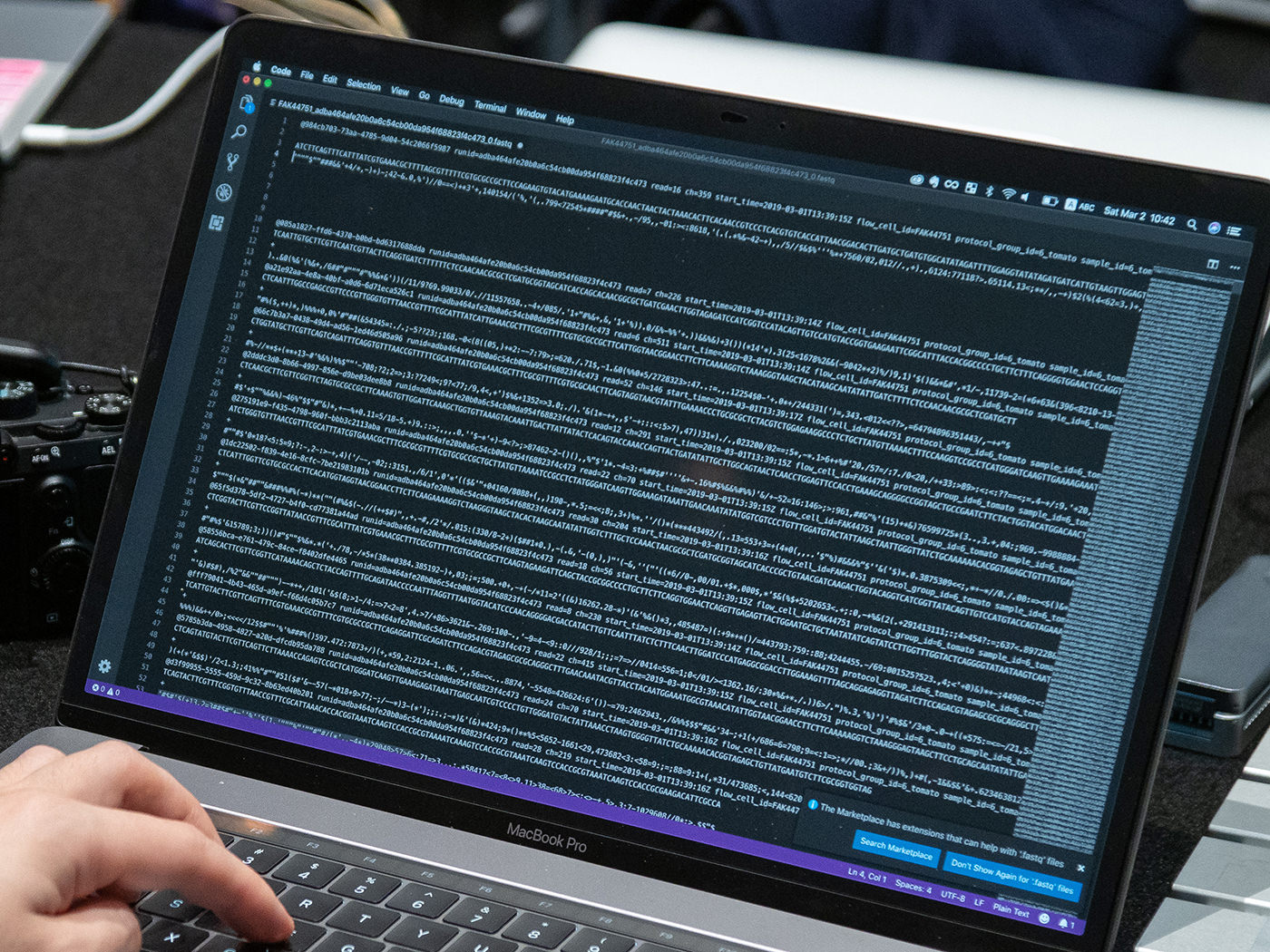 ほとんど宇宙からのメッセージ状態で、初心者には全く理解が追いつかない。
ほとんど宇宙からのメッセージ状態で、初心者には全く理解が追いつかない。
一つ目のリードの4行目、クオリティスコアを部分的に抜き出して見るとこのような内容だ。
" " " " $ " " # # # & & ' + 4 / * , - ) + ) - ; 4 2……
これをアスキーコードの10進数に対応させていくと以下になる。
34 34 34 34 36 34 34 35 35 35 38 38 39 43 52 47 42 44 45 41 43 41 45 59 52 50……
これをさらにクオリティスコアに換算すると次のようになる。
1 1 1 1 3 1 1 2 2 2 5 5 6 10 19 14 9 11 12 8 10 8 12 26 19 17……
クオリティスコア(Q値)の高さは、解読の精度に比例しており、Q値が低いリードは「正確に読めていない」ことを意味する。例えば、Q値「1」を変換式から逆算すると、エラー率は約79%(p≒0.79432)となり、配列の信頼度は約21%しかない。
一方、会場のあちこちでは、多くの参加者から困惑の声が上がっていた。バイオインフォマティクス の心得のない初心者にとって、いささか作業内容が難しすぎたのだ。
まず現状のバイオインフォマティクス は、 Mac のターミナルや Linux上などCUIでの操作が前提となっている。プログラミングの知識がなく、マウスでの操作しかしたことがない人にとっては、ターミナルからコマンドを打つこと自体が非常に高いハードルに感じられる。
 パソコンを前にして、厳しい表情を浮かべる参加者たち。
パソコンを前にして、厳しい表情を浮かべる参加者たち。
加えて、クオリティスコアの難解なルールや解釈についても、わずか数時間のレクチャーでは消化できず、各チームとも苦労している様子が見られた。そのため片山氏や鎌田氏、荻島氏をはじめ、YCAMスタッフも総出で会場を走り回り、サポートに追われていた。
 ファイルの格納場所や記述された内容について各テーブルを説明して回る鎌田氏(後列右)
ファイルの格納場所や記述された内容について各テーブルを説明して回る鎌田氏(後列右) 壇上と会場を行ったり来たりしながら説明する片山氏のアドバイスに聞き入る参加者たち
壇上と会場を行ったり来たりしながら説明する片山氏のアドバイスに聞き入る参加者たち GitHubで共有された片山氏の資料を参考にしながら、リードの解読を進める。
GitHubで共有された片山氏の資料を参考にしながら、リードの解読を進める。
トマトチームのテーブルでも、混乱が起きていた。
「リードの端っこの方は、クオリティスコアが 1 とか 2 ばっかりですね」
「でも右に行くにつれて、だんだん数値は高くなってるね」
「スコア 10 で信頼度は 90%だけど、それって精度として高いの? 低いの?」
「10回に1回は読み間違えるってことだと、けっこう影響は大きいよね」
そんな会話をしていたところ、YCAMディレクターの伊藤氏が登場。丁寧に解説してくれた。
「通常、読み始めと読み終わりに近い部分はクオリティスコアが低く出やすいんです。一方、信頼度 90% という結果をどう評価すべきかは、ケースによって異なるでしょう。例えば、ゲノム医療の世界では、たった1塩基の違いががんや難病といった疾患の発症を左右します。その場合、1割の解読エラーが与える影響は大きいです。だからこそ、同じ遺伝子領域を何度も読み、その結果を重ね合わせることで、配列決定の精度を上げていくんです」
次の工程では、FASTQファイルの配列情報を決定済みの完全ゲノムデータ(リファレンス配列)にマッピング(アライメント)していく。マッピングとは、バラバラに読まれたリードの配列が、リファレンス配列のどの染色体のどの部分の配列に由来するものか調べることだ。
FASTQファイルには、配列の断片が読んだ順に記録されている。リードが正しく読めているかどうか(サンプルの食材のゲノム情報と一致するか)を確かめるには、ゲノムの座標どおりに断片化した配列を正しく並べ直してチェックする必要がある。たとえるならマッピングは、読んだ配列の住所地番を探し出す作業だ。

参照元となるリファレンス配列は、国内外の公共データベースから最適な塩基配列データを入手して構築する。DNA配列データベースとしては、米国国立生物工学情報センター(NCBI)の「GenBank」や欧州バイオインフォマティクス研究所(EMBL-EBI)の「ENA」のほか、日本の国立遺伝学研究所(NIG)が運営する「DDBJ」などが知られている。これらのデータベースは互いに連携しており、いずれかのデータベースに新しく情報が登録されると自動的にそれぞれのデータベースにも共有・反映されるため、生命科学分野の国際的な情報プラットフォームとして重要な役割を果たしている。
今回のワークショップでは、すでに片山氏らが最適なリファレンス配列を作り、フォルダ内に用意してくれていた。なお、配列データはクオリティスコアを含まない「FASTA(ファストエー)」とよばれるファイル形式で記述される。リファレンス配列を自分で構築してみたい人は、片山氏のGitHub に詳しい作成方法が掲載されているので、参考にしてほしい。
https://github.com/ktym/GenomeBento/
マッピングには「BWA(Burrows-Wheeler Alignment)」と呼ばれるツールを使用する。BWAは 200塩基未満のショートリードのマッピングに長く用いられてきたプログラムだが、ロングリード(70bp〜1Mbp まで)向けのアルゴリズムとしてサブコマンドの「bwa mem」も実装されている。今回はワークショップ用のパソコンの中にBWAがインストールされた状態になっていたが、もし自分のパソコンでマッピングしたい場合は、専用サイトからプログラムをダウンロードしてインストールする必要がある。
マッピングの対象となるのは、全ゲノムをシーケンスした配列決定済みの食材①(白菜、ひよこ豆)のデータだ。そこで②と③の食材を選んだ残り4チームも、白菜とひよこ豆のどちらかを選んでマッピングを体験してみることになった。我々トマトチームは、白菜のデータで演習することとした。

今回、MinION の出力する FASTQ ファイルは、 4000リードごとに1つのファイルになるように設定されている。今回は、複数のファイルに分かれて保存されたシーケンスデータをサンプルごとに1つの FASTQ ファイルにまとめてからマッピングを行う。下準備として、ターミナルからcat コマンドを実行し、複数のファイルを1つに繋げておく。
続いて、BWA コマンドでリファレンス配列に対してインデックスを作成する(今回は片山氏らがすでに作成済み)。このインデックスを使ってマッピングアルゴリズム「bwa mem」を実行し、先ほど一つにまとめた白菜(またはひよこ豆)のFASTQファイルをそれぞれのリファレンス配列にマップする。
一方、会場では、難解なマッピングのしくみやBWAコマンドに対する理解が追いつかず、作業につまづくチームが多数見られた。トマトチームでは、京都から参加したエンジニアの池田航成氏、東京から参加した藤原純氏の奮闘で、何とかBWAコマンドの実行に成功。マッピング自体は15秒ほどであっさり完了した。
 トマトチームで解析作業をリードしてくれた池田氏(前列右)と藤原氏(同左)
トマトチームで解析作業をリードしてくれた池田氏(前列右)と藤原氏(同左)
マッピングの結果は「SAM」と呼ばれるファイル形式で保存される。SAMファイルには、シーケンスした1本1本のリードがどの染色体のどこの配列にマップされたかが記載されている。このSAMファイルを圧縮したものは「BAM」ファイルと呼ばれる。
SAMファイル上のマッピング結果を確認するには、ゲノムブラウザを使って内容を視覚化する必要がある。ゲノムブラウザとは、配列情報や遺伝子の位置など、ゲノムに付随する情報(アノテーション)を可視化するツールのことだ。今回は「IGV」と呼ばれるゲノムブラウザを使用する。
各チームの共用パソコンにインストールされたIGVを起動し、選んだ食材(白菜またはひよこ豆)のリファレンス配列(IGVでの作業用にFASTAファイルを変換したもの。拡張子は .genome )を読み込む。プルダウンメニューから、各染色体ごとの DNA 配列が選択できる状態になっていれば、正常に読み込まれた証拠だ。
次に、IGVに内蔵されているツール( igvtools )を使って、先ほどつくったSAMファイルをゲノムの座標順に並べ替える。なぜなら、SAMファイルの配列情報はゲノムの座標どおりではなく、シーケンスされた順に記録されているからだ。並べ替えた SAMファイルは「.sorted.sam」の拡張子がついたファイルとして保存される。
最後に、この「.sorted.sam」ファイルをIGVに改めて読み込むと、マッピング結果を見ることができる。ブラウザの任意の場所をズームすると、リードの断片がリファレンス配列のどこにマップされたのかを視覚的に確認できるしくみになっている。
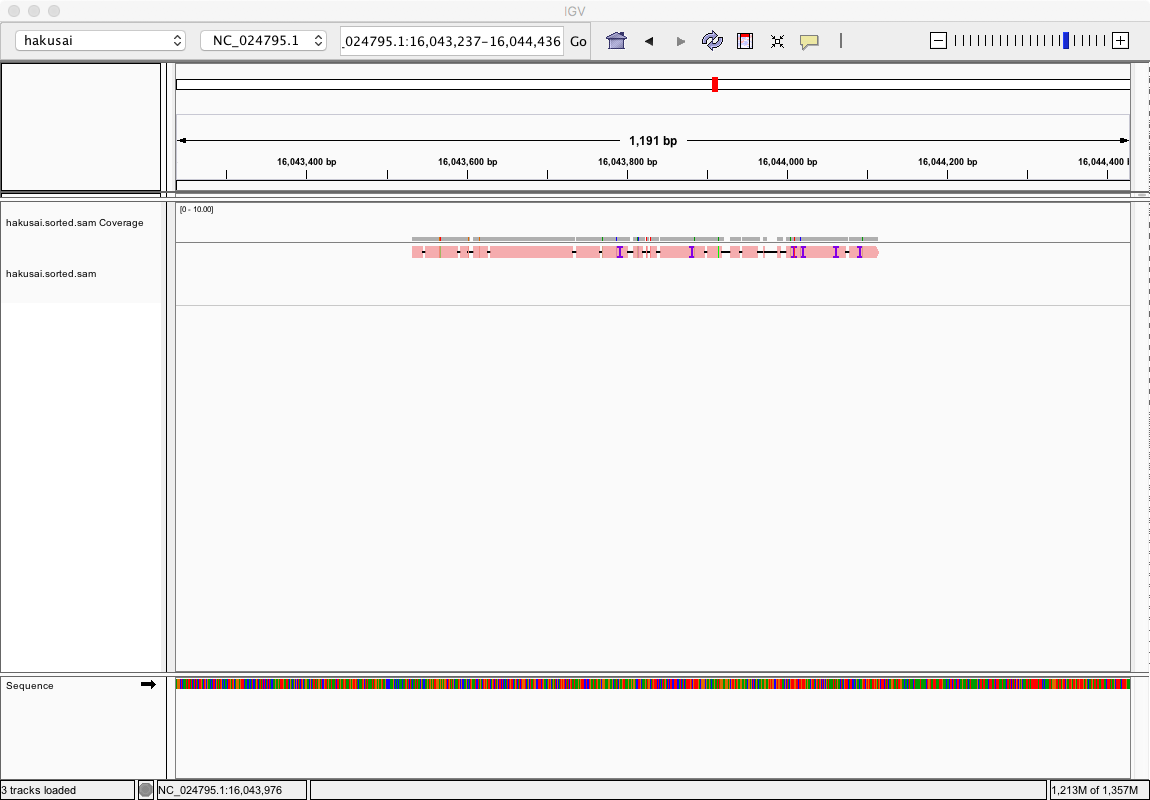 リファレンス配列に一致するリードの断片は赤く表示されている。(片山氏のプレゼン資料より)
リファレンス配列に一致するリードの断片は赤く表示されている。(片山氏のプレゼン資料より)
SAMファイルのゲノム位置情報も参照しながら、ズームやスクロールを繰り返し、マップされたリードを探すと、ようやくそれらしき箇所を発見できた。
「あー、いたいた! あったあった!」
「かわいい〜」
「こっちにもいた! けっこうマップされてる」
そんな歓喜の声が各テーブルから聞こえてくる。無事にリードがマップされていることが確認でき、会場からは大きな拍手が上がった。
 マッピングに成功し、思わずIGVの画面を撮影する参加者たち。
マッピングに成功し、思わずIGVの画面を撮影する参加者たち。
続いて、全ゲノム配列が部分的に決定済み(ドラフトゲノムあり)のサンプル②(にんじん、トマト)、複数の食材が混在する料理サンプル③(炊き込みごはんの具、漬物)から、 rbcL 遺伝子を検出する「植物のメタバーコーディング解析」に挑戦してみる。rbcL は葉緑体ゲノムに含まれる遺伝子で、植物種ごとに異なる場合が多いため、DNA による植物種同定のバーコードのひとつとして活用されているという。
基本的なプロトコルは、先ほどのリードマッピングと同じだ。PCR で増幅した rbcL 遺伝子領域の リードデータ(FASTQファイル)を、BWAでリファレンス配列にマップし、SAMファイルを作成する。参照元となるリファレンス配列は、下記の論文にある8万7千種の植物から集められた rbcL 遺伝子の配列データセットを元に、片山氏らがあらかじめ用意してくれた特別なデータベースを使用した。
“An rbcL reference library to aid in the identification of plant species mixtures by DNA metabarcoding.” Bell KL, Loeffler VM, Brosi BJ.
Appl Plant Sci. 2017 Mar 10;5(3). pii: apps.1600110. doi: 10.3732/apps.1600110. eCollection 2017 Mar.
https://www.ncbi.nlm.nih.gov/pubmed/28337390
リードがリファレンス配列上で、読んだサンプルに含まれる食材と同じ生物種の rbcL 配列にマップされていれば、実験は成功といえる。今回はゲノムブラウザではなく、片山氏らが用意してくれた簡単なスクリプト(check.sh)を使ってSAMファイルの内容を検証する。
check.sh は、SAMファイルのデータを元にリファレンス配列に、マップされたリードの本数を生物種ごとにカウントしてくれる。ターミナルからコマンドを実行すると、似ている配列が多い順にソートして結果が表示された。
 似ている配列を検索した結果の中に「トマト」が出てこない……。
似ている配列を検索した結果の中に「トマト」が出てこない……。
トマトチームでは、最も似ている配列が多かったのは「じゃがいも」という結果となった(全リードのうち749本が一致)。下位まで結果をスクロールしても、トマトの「ト」の字もない。チームに落胆ムードが広がる中、片山氏から次のような助け舟が入った。
「じゃがいももトマトもナス科だから、近いと言えば近いですね。また、結果の上位に表示されている『Solanum lycopersicum』はトマトの学名なので、わりとうまく読めていたと思います」
一通り解析が終わったところで、片山氏からウェブブラウザ上で配列の相同性検索ができるツールについての紹介もあった。NCBI が運営する「BLAST(Basic Local Alignment Search Tool)」は、任意の配列データ(問い合わせ配列)に類似した配列をデータベース中から検索、収集できる便利なツールだという。
https://blast.ncbi.nlm.nih.gov/Blast.cgi
BLASTのサイト上で、テキストボックスに問い合わせ配列のテキストデータを直接入力、またはFASTA形式の配列データをアップロードして実行ボタンを押すと、配列が類似した生物種の候補リストが出力される。試しに、FASTQファイルからコピーしたトマトの配列データの一部を貼り付けてBLAST検索したところ、3件がヒット。いずれもトマトの学名を持つ配列との類似性が確認できた。
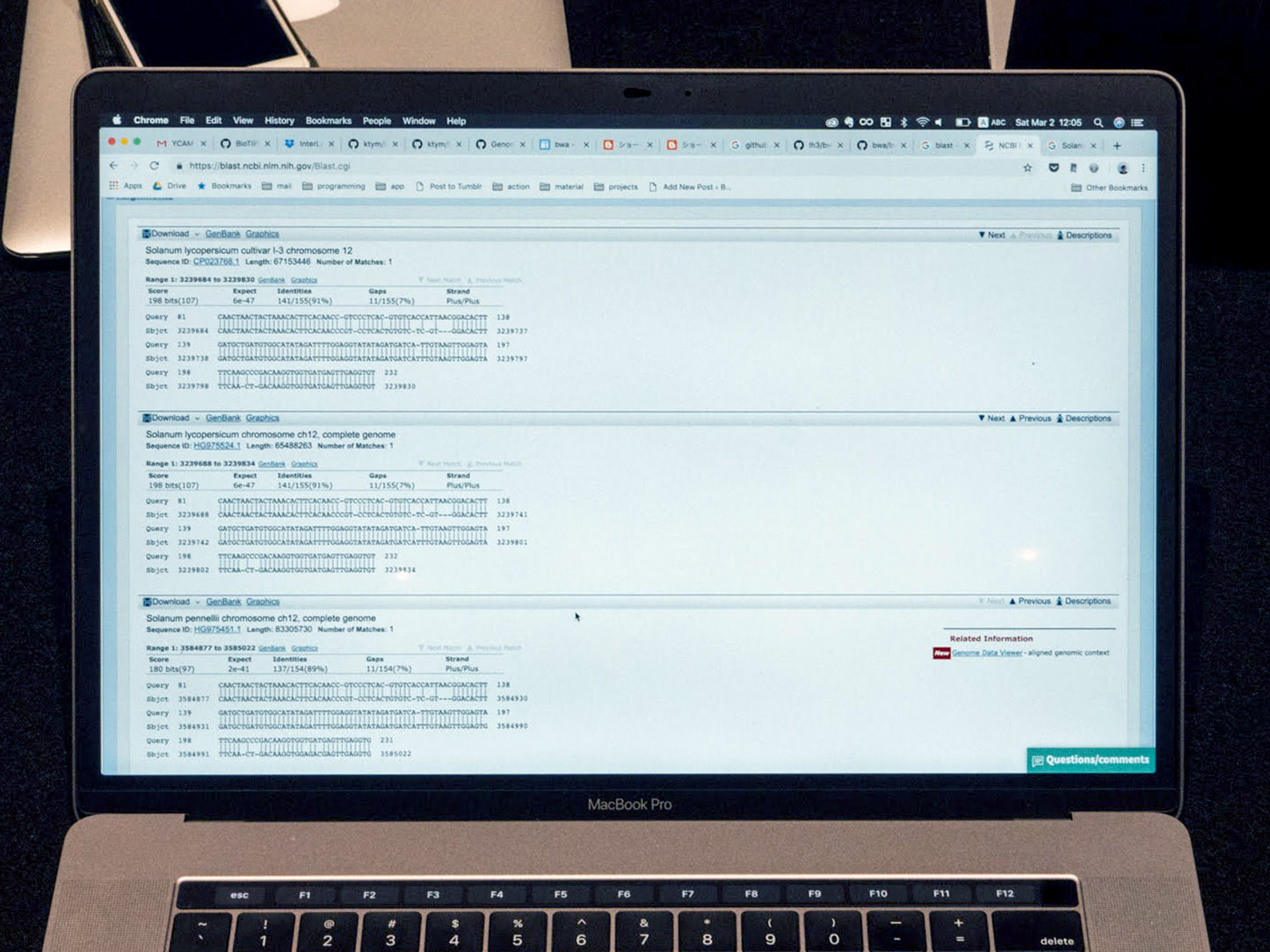 BWAを使ったマッピングでは「じゃがいも」判定されたが、NCBI BLASTでは「トマト」の確率が高まった。
BWAを使ったマッピングでは「じゃがいも」判定されたが、NCBI BLASTでは「トマト」の確率が高まった。
NCBI BLASTのように便利なツールがあるのなら、わざわざコマンドを叩く必要はない気もする。しかし、大量のリードデータをすべてブラウザ上で解析する場合は大きな負荷がかかるため、今回はあえてローカルにデータベースを構築して解析する手法を取ったようだ。
今回体験したゲノムマッピングや植物のメタバーコーディング解析は、ゲノム解析の初歩の初歩にすぎない。実際の現場では、この段階を起点にマップされたリードがどの遺伝子に該当するのか分析したり、リファレンス配列との比較から変異の有無を調べるなどの研究が始まるそうだ。
演習後、参加チームのメンバーに感想をたずねると、さまざまな声が聞かれた。
「今後パーソナル・バイオテクノロジーが普及していくためには、FASTQファイルからマッピングまでを直感的に操作できる、誰もが扱えるユーザーインターフェースの開発が必須だろう。現状は、プログラミングの素養がないと厳しい印象」(先述のエンジニア・池田氏)
「今のバイオは、コンピュータで言えばMS-DOSの時代。その後、PCの世界に Windows が登場したように、バイオの分野にもこれからどんどん便利なツールが登場してくるのでは」(ファブラボ浜松/TAKE-SPACE 代表の竹村真人氏)
1970年代、新しく登場した8ビットマイクロプロセッサーを使い、ガレージやバックヤードで試行錯誤を重ねた人々がいた。彼らの好奇心と情熱は、やがてパーソナル・コンピュータの普及という大きなムーブメントにつながった。今回のワークショップで、バックグラウンドの異なる人々が額を寄せ合い、バイオという新たな技術に奮闘する光景は、まるでPC黎明期の興奮や感動を追体験しているようだった。今まさに開花しつつあるパーソナル・バイオテクノロジーの現場に当事者として立ち会えたことは、とても幸運なことだと感じた。
 いつかこの光景が、パーソナル・バイオテクノロジー黎明期の貴重なワンシーンとなる日が来るかもしれない。
いつかこの光景が、パーソナル・バイオテクノロジー黎明期の貴重なワンシーンとなる日が来るかもしれない。ゲノム解析の演習後、ランチタイムへ。2日目のゲノム弁当も「ベジタブル喫茶 ToyToy」プロデュースによるものだ。今日はゲノム解読済みの食材と調味料だけを使った正真正銘のゲノム弁当。ラインナップには、先ほど解読に挑戦した白菜のお漬物や炊き込みご飯も(!)。ATGCの塩基配列を想像しながら、一つ一つのメニューを味わった。
 鮭と白菜のペンネ、キャベツと青梗菜のサラダ、鶏肉の味噌焼き、春雨と豚肉炒めなど。シーケンサーの進歩がなければ、彩り豊かなお弁当は作れなかったと思うと感慨深い。
鮭と白菜のペンネ、キャベツと青梗菜のサラダ、鶏肉の味噌焼き、春雨と豚肉炒めなど。シーケンサーの進歩がなければ、彩り豊かなお弁当は作れなかったと思うと感慨深い。
制作を担当した「ベジタブル喫茶 ToyToy」の村田敦氏によれば、ゲノム弁当に使う食材は、このデータベースに掲載されたものから採用しているという。
データベースは、片山氏らが論文検索などで最新のゲノム解読情報をチェックし、新しい食材を随時追加しているそうだ。村田氏は「しょうがとニンニクが追加されれば、かなり味付けの幅が広がるので、1日も早い解読を期待しています」と話した。
 「食材の縛りがきつい中、これだけ美味しいお弁当がつくれたのは ToyToy さんのおかげ」と片山氏。
「食材の縛りがきつい中、これだけ美味しいお弁当がつくれたのは ToyToy さんのおかげ」と片山氏。
午後の部は、YCAMの津田氏より、ゲノムの「書き」に関する説明が行われた。「書き」については実際の演習ではなく、レクチャーを中心に展開される。
「ゲノムの『書き』に相当するのは、いわゆる合成生物学と呼ばれる分野です。従来の生物学に工学(エンジニアリング)の概念を組み合わせた学問領域と言えます」(津田氏)
 1日目に続きプレゼンテーションを行ったYCAMの津田氏
1日目に続きプレゼンテーションを行ったYCAMの津田氏
合成生物学では、工学のデザイン(設計)・ビルド(合成)・テスト(検証)というサイクルに沿って、あたらしい生物システムを構築することを通じて、生命への理解を深めていくことを目指している。従来の遺伝子導入の技術や、ゲノム編集技術などを応用したさまざまな研究が行われているという。
「代表的な事例では、設計したDNAをプラスミドと呼ばれる環状DNAに組み込み、それを大腸菌や酵母などに導入して大量培養させることで目的のDNAを増幅し、狙った機能を実現させる研究があります。こうした技術を使って、バイオ燃料や医薬品の原料を大腸菌につくったり、有毒物質を感知したりする応用研究がすすめられています」(津田氏)

近年では、生命そのものをつくり出す研究も進められている。2016年には、生命として機能する最小ゲノムを持つ人工細菌(ミニマル・セル)が発表され、話題となった。
こうした動きを背景に、合成生物学の発展を目的とした世界規模のコンペも開催されている。DAY1 にフィリップ・ボーイング氏も言及した「国際合成生物学大会(iGEM)」だ。
「iGEM では、参加チームが標準化されたDNAパーツ『バイオブリック(BioBrick)』を組み合わせ、新たな生物システムのアイデアを競い合います。一方で、 iGEM では合成生物学を取り巻くバイオセーフティ(安全性)や生命倫理といった課題を非常に重視していて、厳格なルールのもと運営されています」(津田氏)
津田氏は、自身が監訳に携わり、2018年11月に刊行された書籍『バイオビルダー ―― 合成生物学をはじめよう』についても言及。本書は「国際合成生物学大会(iGEM)」のフレームワークに沿って構成されており、合成生物学の基礎から実験演習のプロトコル、生命倫理までを一通り学べる入門書となっている。これから「DIYバイオ」に取り組む人々にとっては、必読の一冊と言えそうだ。
 『バイオビルダー――合成生物学をはじめよう』(オライリー・ジャパン/2018年11月発売)
『バイオビルダー――合成生物学をはじめよう』(オライリー・ジャパン/2018年11月発売)
続いて、米国ニューヨークを拠点にDIYバイオロジスト/ モレキュラー・フローリスト として活動するセバスチャン・コシオバ氏の講義が始まった。
コシオバ氏は、大学で生物学を学んだ後に中退し、以降は独学で遺伝子工学や合成生物学を学んだという。現在は、DIYバイオに取り組む有志のメンバーとともに「Binomica Labs」という研究グループを主宰。主に植物(花)を対象とした研究を続ける一方、その成果をオープンソースで公開したり、市民向けにワークショップを開くなど、DIYバイオのエヴァンジェリストとしても活動している。
 幼い頃から美しい草花や植物に魅了され、「生物の進化の秘密を知りたい」という思いからDIYバイオに取り組み始めたコシオバ氏。
幼い頃から美しい草花や植物に魅了され、「生物の進化の秘密を知りたい」という思いからDIYバイオに取り組み始めたコシオバ氏。
彼のラボは、マンハッタンのスカイラインを見渡す自宅アパートの一角にある。寝室の一つを改造し、必要な機材は自分で揃えた。米国のバイオセーフティレベル1(BSL1)の基準もクリア済みだという。バイオセーフティレベルとは、細菌やウイルスを扱う実験室に対し、WHO(世界保健機関)の指針に沿って各国が独自に定める安全基準のことだ。コシオバ氏は、バイオラボのある家で母親と”ふつうに”暮らしながら研究を行う生活を、もう15年も続けている。
 自宅の一角に設置したバイオラボの様子。
自宅の一角に設置したバイオラボの様子。
コシオバ氏は、このラボで取り組む「読み」のプロジェクトとして、まずオキサリス(カタバミ)のDNA解読についての研究を紹介した。
「オキサリスは世界中のあちこちで見られる雑草です。私は各地域のオキサリスの大きさや色にさまざまなバリエーションがあることに注目しました。なぜこの植物が様々な場所に生息し、多様な遺伝子を持ちながら、同じ種に属すことができるのか? 地域ごとの形態の違いについて調べれば、その進化の謎に迫ることができると考えたのです」(コシオバ氏)
 オキサリスは日本ではカタバミと呼ばれ、庭や道路脇などで簡単に見つけることができる。
オキサリスは日本ではカタバミと呼ばれ、庭や道路脇などで簡単に見つけることができる。
研究の過程では、12歳の少女が快挙を成し遂げた。彼女は10歳のとき、コシオバ氏の開催するワークショップに参加。そこでオキサリスの栄養源について疑問を持ち、自らその生態を調べ始めた。
「100本のオキサリスに、混合濃度を変えた5つの肥料を与えて生育状況を調べる実験を3回繰り返しました。彼女は葉の数や水分量を詳細に記録しながら、カメラを設置してタイムラプスで撮影。その様子を毎日ツイッターに公開していました。得られたデータについては、私が統計学を教え、彼女自身が分析を行いました」
彼女は研究成果をサイエンスコンテストで発表し、見事に優勝。オッタチカタバミの必須栄養素を解明したのは、この12歳の少女が世界初だという。
彼女の実験で得られた栄養素の知見は、コシオバ氏が主導するオキサリスDNA解読プロジェクトにも大きく寄与した。その後、コシオバ氏の研究チームはMinIONを使用したシーケンシングを行い、5億ほどのリードデータを取得し、そのうちの約20万リードが一つにつき200万塩基対もあったという。
専門知識や学位を持たないアマチュアでも、個人のラボで世界レベルの研究ができる――。彼らのケースは、イノベーションを加速するDIYバイオの可能性を示す好例だろう。
次にコシオバ氏が「書き」のプロジェクトとして紹介したのは、青色のイチゴをつくる試みだ。コシオバ氏はサントリーが開発して話題となった「青いバラ」をスライドで見せながら、その色が実際には青ではなくラベンダーに見えると指摘。誰が見ても青色だと思える植物の開発を、どうせならバラではなくイチゴで試してみようと考えたという。
「それは言うなれば、『チャーリーとチョコレート工場』に登場する架空のイチゴ(スノッズベリー)のようなものです」
 誰が見ても「青色」だと思える植物をつくりたい、と語るコシオバ氏。
誰が見ても「青色」だと思える植物をつくりたい、と語るコシオバ氏。
コシオバ氏によれば、青色を発現するタンパク質は海に12種類存在するという。彼はその中から、熱帯地域に生息するアサリに含まれるタンパク質に注目。このタンパク質をつくる遺伝子を、土壌細菌の一種であるアグロバクテリウムに組み込むことにした。
コシオバ氏は、このタンパク質を つくる遺伝子をバクテリアに組み込み、37世代にわたり培養。つい先日、青色のタンパク質を発現するバクテリアの生育に成功したという。
その遺伝子を、土壌細菌の一種であるアグロバクテリウムに組み込むことにした。 アグロバクテリウムは、植物に感染することで自分の遺伝子を植物に導入する性質を持ち、遺伝子の運び屋(ベクター)として盛んに活用されている。つまり、青色となるタンパク質を発現する遺伝子をアグロバクテリウムに組み込み、目的の植物に導入すれば、青色の花や実をつくることも可能なわけだ。
 遺伝子導入したバクテリアから抽出した青色のタンパク質。その名も「Bino Blue (Binomica Blue)」
遺伝子導入したバクテリアから抽出した青色のタンパク質。その名も「Bino Blue (Binomica Blue)」
「青色をつくるためのDNAは獲得できたので、現在はこのアグロバクテリウムでペチュニアに遺伝子を組み込み、青色の花を咲かせる実験に挑んでいます。植物の中で安定的に青いタンパク質を発現させられる仕組みを確立できれば、次は本命の『青いスノッズベリー』の開発に取り組む予定です」
続いてコシオバ氏は、自身が少しコンセプチュアルなプロジェクトであると語る「インフィニット・ディスカバリー・マシン(infinite discovery system)」について紹介した。
「すべての生命体は、淘汰を繰り返しながらタンパク質を次世代に継承し、進化してきました。それは、タンパク質の発現が進化の過程で生み出された秩序に則って行われていることを意味します。しかし、自然界のランダムなDNA配列から”偶然”、生成されるタンパク質は本当にないのでしょうか。そんな素朴な疑問をきっかけに、このプロジェクトは始まりました」
コシオバ氏らはまず、ランダムなDNA配列からタンパク質を生み出すプログラムをつくり、それらの配列を地球上の既知のタンパク質と比較するシミュレーションを行った。さらに、実際にタンパク質として発現させた場合、どのような毒性や危険性があるかの評価も試みた。
「その結果、”偶然”によって生み出されたタンパク質は、自然界のどのタンパク質とも類似しておらず、毒性や危険性についても不明なままでした。つまり、”わからないものはわからない”。私たちは地球上に存在しない、未知なものを分析することはできないからです。しかし、そんな謎だらけのタンパク質でも、バクテリアに導入すれば発現させることができてしまう。それは生命倫理の観点では、非常にグレーであると言えます」
コシオバ氏は、倫理的な問題に留意しながら実験を続けた。まず、オキサリスのDNAを単離し、酵素を使ってDNAを1塩基ずつバラバラに切断。DNA同士をつなぐ”のり”の機能を持つDNAリガーゼを用いて、ランダムにつなぎなおした。次に、このDNA鎖をプラスミドに組み込むために、特定の塩基配列のみを切断する制限酵素を使い、つないだ配列を再び切断。これらの断片を組み込んだプラスミドをバクテリアに導入し、ランダムに生成した”地球上に存在しない”タンパク質の発現に成功したという。

「私たちはできたタンパク質をフリーの構造解析ツールを使って確認しました。その構造は、やはり地球上に存在するいかなるタンパク質とも類似性がないと判明しました。この発見は、触媒としての活用や新薬開発など無限の可能性を持つと同時に、大きな倫理的な問題も抱えていました。もし存在しないはずのタンパク質が現存する生命体と関わったら、どのような影響が出るのでしょうか。私たちは生命倫理の専門家に意見を求めました」
しかし、彼らの答えは総じて「やってみなければわからない」というものだった、とコシオバ氏。この未知なるタンパク質が、農業や人命に脅威をもたらす可能性はゼロではない。コシオバ氏は、研究がもたらす影響を慎重にリスクアセスメントするため、このプロジェクトを完全な公開プロジェクトとして続けている。研究に関するデータセットは全てオープンソースとして公開され、誰もがアクセスできる状態にしているそうだ。
「私が母親のアパートの一室で研究を続けているように、今は誰もがサイエンティストになれる時代。DIYバイオの世界では、コンセプチュアルなものだけでなく、社会に有用なプロジェクトも数多く生まれている。私はこれからもDIYバイオを通じて科学の発展に貢献していきたいと思っています」
“Small Thoughtful Science” セバスチャン・コシオバ氏コシオバ氏のプレゼンテーションに続いて、早稲田大学理工学術院教授の岩崎秀雄氏が登壇。岩崎氏は、光合成を行う細菌「シアノバクテリア」に関する研究活動を行う一方で、バイオアーティストとしても活動している。
 早稲田大学理工学術院教授・岩崎秀雄氏。生命美学プラットフォーム「metaPhorest」を主宰する。
早稲田大学理工学術院教授・岩崎秀雄氏。生命美学プラットフォーム「metaPhorest」を主宰する。
彼はまず、DIYバイオ(パーソナル・バイオテクノロジー)の応用可能性を検討する上で欠かせない生命倫理上の課題や規制のあり方について、いくつかの観点から整理した。
DIYバイオにおいて、何よりも重要なのはやはり「安全性」だ。岩崎氏は「生態系の撹乱、人体への副作用などには最大限の配慮が必要」と強調する。
「遺伝子を組み換えた生物が野外に出た場合、生態系や人体にどのような影響を与えるかは未知数です。その危険性をいち早く予測し、利用にブレーキをかけたのは、他ならぬ科学者たち自身でした。彼らは1973年に遺伝子組み換え技術が登場すると、すぐさま安全に研究を進めるためのルールづくりに取り組みました。1975年に行われた国際会議の内容は、遺伝子改変を伴う研究を規定する各国の法律やガイドラインに大きな影響を与えています」
これを受けて1979年、日本でも当時の文部省と科学技術庁によって「組換えDNA実験指針」が取り決められた。その後、2004年に「遺伝子組換え生物等の使用等の規制による生物の多様性の確保に関する法律」が施行され、指針から法律へと格上げされた。これは通称「カルタヘナ法」と呼ばれ、明確な罰則規定も設けられている。現状、企業や大学が遺伝子の改変を伴う実験を行う場合は、カルタヘナ法の遵守が求められ、実験内容の事前審査や各関係省庁からの認可などが必要となっている。
 岩崎氏のプレゼン資料より
岩崎氏のプレゼン資料より
岩崎氏は、生命倫理をめぐる二点目の課題として、この「ルール・規定のあり方」を挙げる。なぜなら、近年では技術の進化に法整備が追いつかず、法律でカバーできないグレーな事例が増えてきているためだ。
「例えばゲノム編集技術の場合、自然界でも起こりうる塩基の置換などについては現行のカルタヘナ法に抵触しないケースがあります。また、個人が自身の体内に自前の遺伝子を導入するようなケースでは、規制の対象外となる可能性もある。このようにグレーな領域が広がると、責任の所在が不明確となる問題も浮上してきます。バイオテクノロジーのオープン化と責任をどのように両立していくか。これはDIYバイオを進める上で、大きな課題です」
 「どのように生命倫理のルールがつくられてきたかを知ることも非常に重要」と語る岩崎氏。
「どのように生命倫理のルールがつくられてきたかを知ることも非常に重要」と語る岩崎氏。
さらに岩崎氏は、情報やメディアに関する課題についても触れた。DIYバイオにおいては、アカデミズムのように研究成果を論文にまとめ、検証可能な形で発表するという慣行がなく、しばしば情報公開の不十分さやフェイクの混在リスクなどが指摘されている。岩崎氏は「スペキュレーション(思索)とファクト(事実)が混在し、判断できなくなることが最大の問題」と強調する。
「生命や身体を主題として扱う以上、そこには社会的責任が伴います。たとえそれがスペキュラティブなアート作品であったとしても、事実確認や第三者による検証が可能なものであるべきでしょう。それはDIYバイオを扱う者が最低限、身につけるべきリテラシーだと思います」
 生命をめぐる多様な視点について解説する岩崎氏
生命をめぐる多様な視点について解説する岩崎氏
このように、DIYバイオにはまだまだ検討すべき倫理上の課題がある。しかし一方で、そこには大きな可能性も秘められている。中でも最も期待がかかるのは、医科学などにおけるボトムアップ型イノベーションだが、岩崎氏は「産業界やビジネスの文脈に取り込まれない、DIYが持つ本来の文化的価値も重視されるべきではないか」と提言する。
「もともとDIYの文化とは、自ら手を動かし、工夫することで身の回りの生活を豊かにしようというもの。バイオテクノロジーはその一つの手段であって、目的ではない。実際にDIYバイオを実践してみれば、期待通りの結果を得るのは難しく、生物のDNAを扱うことがそれほど簡単でないことはすぐにわかる。むしろ、その試行錯誤のプロセスを通じて、生命の本質や自分自身の存在を問い直し、理解することに大きな価値がある。私はそう考えています」
岩崎氏自身も、2012年に自宅アトリエ内にバイオラボを構え、DIYバイオを実践している。2014年には、培養したシアノバクテリアがコロニーを形成し、やがて渦を形成しながら遷移していく現象を観察した。世界で初めてかもしれないという。
 岩崎氏が私設したバイオラボ。機材の多くはオークションサイトで購入したものだという。
岩崎氏が私設したバイオラボ。機材の多くはオークションサイトで購入したものだという。
「生物の群れを観察すると、初めはランダムに動いていた個体が次第に周囲の動きに合わせて一方向に移動しはじめるなど、自発的に秩序立ったパターンや構造を形成する現象がよく見られます。今朝のコンタクトゴンゾさんのワークショップも、この現象を想起させるような内容でした。現在は、こうした現象を数理モデルによって解析し、生物の秩序形成メカニズムを解明する取り組みを続けています。重要なのは、このような発見が自宅のローテクな顕微鏡でもできるということ。それこそが、DIYバイオが秘める最も大きな可能性だと思います」
最後に岩崎氏は、DIY バイオのアートへの応用とその意義について語った。冒頭で紹介したとおり、彼は生物学者として「生命」の研究を続ける一方、バイオアーティストとして「生命」を探求する活動も行っている。2007年からは、生命に関する美学的な研究・制作を行うプラットフォーム「metaPhorest(メタフォレスト)」を主宰。「生命とは何か」という命題を、生命科学のみならず人文学の視点からも照射し、研究するオープンな場を構築している。
 現在は研究者をはじめアーティストやデザイナーなど多様なバックグラウンドを持つメンバーが10名ほど在籍する。
現在は研究者をはじめアーティストやデザイナーなど多様なバックグラウンドを持つメンバーが10名ほど在籍する。
岩崎氏は、生命に対してアートとサイエンスの両面からアプローチする理由を「科学が対象にする生命と、私たちの日常の中で語られる命を地続きにするため」と語る。
「自然科学では、生命を研究対象の中に宿る観測可能な現象として捉えます。一方、アートや哲学では、観察者との関係性の中に宿る体験的なものとして語られる。そのような情動的な生命観は、科学が極力しりぞけてきたものでもある。つまり、科学の側面から見ているだけでは、こぼれ落ちてしまう生命観があるわけです。科学が扱う現象としての生命と、私たちが持っている“いのち”の感覚を、どう接続していくか。そう考えたとき、アートという枠組みを使って語ることが有用だと考えたのです」
科学とアートそれぞれが持つ生命観は、二項対立的に見えて、実際は「互いが互いを包摂し合うメビウスの輪のような関係にある」と岩崎氏は言う。
「生命について考えることは、このメビウスの輪をどう生きていくか、という問いに重なる。多様な生命観のあいだを行き来しながら、生命に思いめぐらせ、理解しようとする態度こそが本質ではないか、と。私にとってバイオアートは、それを実践するためのプラットフォームなのです」
 metaPhorest からは野心的なバイオアート作品が多数生まれている。
metaPhorest からは野心的なバイオアート作品が多数生まれている。
岩崎氏はこれまで取り組んできたバイオアートのプロジェクトを紹介した。その一つが、2013年に発表した『Culturing <Paper>cut』だ。これはシアノバクテリアに関する科学論文を、観察の対象であるバクテリア自身がハッキングし、その立ち位置を反転させるコンセプトのもとに生まれた作品だ。
「科学論文には客観性が求められる一方で、生物学の論文には主観的な記述も多く含まれています。そこで論文の紙面から主観的な表現を切り抜いて培地上に設置し、切り取られた部分にシアノバクテリアを植菌しました。ゆっくりと成長していくバクテリアは、科学者の主観を凌駕しながら、ジェネラティブに新しい模様を描いていく。科学的行為と芸術的表現をメビウスの輪のようにつなぐことを意図した作品です」
 岩崎秀雄『Culturing <Paper>cut』(2013年ー)
岩崎秀雄『Culturing <Paper>cut』(2013年ー)
もう一つの作品は、2016年の茨城県北芸術祭に出展した人工細胞を慰霊するプロジェクト『aPrayer まだ見ぬつくられしものたちの慰霊』だ。生体の機能を再現するため人工的に合成された細胞を、私たちは一つの“いのち”として認めることができるだろうか。そんな人工細胞の生命性を、慰霊という儀式によって逆照射する試みといえる。
「慰霊という行為は、情動的な生命観の究極の姿であり、自然科学が定義する生命像の極北にあるストーリーでもあります。作品化にあたっては、10人以上の人工細胞研究者から人工細胞の“亡骸”を集めてガラスの壺に納めました。地元の方々の協力のもと、埋葬するための石碑を建て、実際に慰霊祭も行いました」
 岩崎秀雄 + metaPhorest『aPrayer まだ見ぬ つくられしものたちの慰霊』より「人工細胞+人工生命之塚」(2016年)
岩崎秀雄 + metaPhorest『aPrayer まだ見ぬ つくられしものたちの慰霊』より「人工細胞+人工生命之塚」(2016年)
その過程では、人工細胞の研究者たちにインタビューも行ったという。彼らの口から紡がれる言葉には、合理的思考と情動のはざまを揺れ動く生命観が浮き彫りにされ、「非常に興味深い言説を得ることができた」と岩崎氏。
「研究者の一人は、いつでも再現可能な人工細胞はそもそも死なないのではないか、と語りました。それはつまり、生命はつくれても死はつくることができない、ということ。非常に重要な視点ですが、科学的規範に照らすと、このような考察は切り捨てられてしまう。バイオアートは、このように人々の中に無自覚に漂う多様な生命観をすくい上げ、可視化していく役割を果たす。私はそう考えています」
「バイオアートとDIYバイオ:Alternativeな生命探究の形を巡って」 岩崎秀雄氏岩崎氏から生命をめぐる新たな視座を得たところで、会田大也氏によるワークショップが行われた。当時、2019年の8月から10月にかけて開催された「あいちトリエンナーレ」でもキュレーターを務めていた会田氏は、YCAMで2003年の開館から11年間にわたりミュージアムエデュケーターとして勤務。ユニークなワークショップを多数開発してきた経歴を持つ。そんな彼が今回用意したのは、自らの思考を整理し、的確に表現する言語化のためのトレーニングだ。
 オリジナルワークショップや教育コンテンツの開発に携わるほか、キュレーターとしても活躍する会田大也氏。
オリジナルワークショップや教育コンテンツの開発に携わるほか、キュレーターとしても活躍する会田大也氏。
「DIYバイオに対する倫理的判断は、社会状況や時代、文化によって変化するものです。このように絶対の正解を持たないテーマについて有意義な議論を重ねるには、内なる思考の解像度を上げ、ディテールまでていねいに言語化する姿勢が重要になります」
そう語る会田氏が参加者に与えたお題は、2人1組で美術鑑賞を行う「ブラインド鑑賞」だ。具体的には、1人に目を閉じてもらい、もう1人が正面のスクリーンに映し出された絵画作品を言葉で説明するという内容だ。
 美術作品を言語だけで鑑賞するというユニークな内容は会田氏が開発した。
美術作品を言語だけで鑑賞するというユニークな内容は会田氏が開発した。
「美術館の展示などで、来場者が一つの作品を鑑賞する時間は、平均して約10秒だと言われています。でも10秒間、鑑賞した後に作品のディテールについて尋ねても、多くの人が覚えていない。つまり人間は、私たちが思っている以上に、目の前にある情報を見ていないわけです。今回のワークショップでは、鑑賞という行為を言語による情報伝達に置き換え、表現力や想像力で互いに情報を補いながら絵画を理解することにチャレンジします」
さっそく各テーブルに2組4名ずつ座り、ワークがスタートした。チームを組んだのは、先述のエンジニア・池田航成氏。制限時間は10分。先攻は池田氏が務め、筆者は目を閉じて聞く側に回った。スクリーンに絵画が映し出されると、池田氏はまず絵画全体の概要を説明する。
池田「畑に夫婦がいて、農作業している。場所はヨーロッパのようですね」
筆者「畑は何色? 農作業は具体的に何をしていますか?」
池田「茶色ですね。球根みたいなものを植えているように見えますね」
筆者「人間は何人いますか? ほかに生き物は?」
池田「人間は2人。腰をかがめるように向き合っています。あとは遠くに牛のようなものが見えます。いや、ロバかもしれないな……」
説明する側は絵画の中に描かれた要素を細部にわたって説明し、聞く側はその情報を脳内で絵画に再構築しながら足りない情報を質問する。各テーブルで、そんなコミュニケーションの応酬が活発に行われていた。
 制限時間いっぱいまで言葉を尽くして絵画の情報を伝えようとする姿があちこちで見られた。
制限時間いっぱいまで言葉を尽くして絵画の情報を伝えようとする姿があちこちで見られた。
制限時間になったので目を開けて実際の絵画を見てみると、想像していたものとだいぶ違っていて驚いた。正解は、西洋絵画の巨匠ジャン=フランソワ・ミレーによる『馬鈴薯の植え』(1861年)。そこで気付いたのは、無意識に脳内で別の作品『落穂拾い』をイメージしていたことだ。池田氏から伝えられた情報と自分の頭の中にある既知の情報を紐づけて、『落穂拾い』にちがいないと勝手に解釈していたのだ。
 ジャン=フランソワ・ミレー『馬鈴薯植え』(1861年頃)。有名な作品なのでワークでは左右反転して表示された。
ジャン=フランソワ・ミレー『馬鈴薯植え』(1861年頃)。有名な作品なのでワークでは左右反転して表示された。
何しろ視覚情報が言語情報に解体されているので、それらを絵画として再構築する際にどうしても自らの知識や経験がバイアスとなってしまう。池田氏は誤解を生まないよう、画面を4つのグリッドに分割してそれぞれの要素の位置関係を明確にしてくれていたにもかかわらず、だ。そこに「言語による鑑賞」の難しさがある。強固な先入観があると認知が歪むという事実を、身を以て体験することになった。
続いては攻守を入れ替え、2つ目の作品の鑑賞に移る。今回のお題はイタリア未来派の画家ジャコモ・バッラの『鎖に繋がれた犬のダイナミズム』(1912年)。ダックスフントを散歩させる黒衣の婦人の足元を、連続写真のように描いた作品だ。

しかし、このような抽象的な表現を言葉だけで説明するのは至難の技だ。そもそも当初は、この絵が何を描いたものなのか説明する筆者自身が理解できていなかった(婦人がゾウガメに見えていた)。風景画と比べて情報が少なく、トリミングの範囲やアングルの説明にも苦戦した。
終了後の振り返りでは、伝わりやすい説明の仕方について共有した。チームを組んだ池田氏の指摘で、印象に残ったのは数値化と具体化の重要性だ。
「数字は誰にとっても同じものなので、伝える過程で誤解が生まれにくい。“人物の大きさは縦方向に対して60%ぐらいの大きさ”のように、できるだけ数値化すると伝わりやすいと感じました。また、憶測や思い込みを排除して前提を解体・再構築し、面倒くさがらずにディテールを伝え合うことも大事ですね」(池田氏)
 ワークショップでの気づきを会場に共有する池田氏
ワークショップでの気づきを会場に共有する池田氏
今回のワークショップでは、同じ対象を見ていても、その体験をどう受け止め、どう解釈するかはそれぞれ違うということを改めて実感することができた。「同じ現実に対して人それぞれの視点がある、そこを出発点にして対話を積み上げていくことが大事」と会田氏は言う。
「そこに何が描かれているのかという事実、そして作品をどのように受け止めたかという解釈を、自分なりの言葉でつなぎとめる。それを他者と共有することで、初めてコミュニケーションが成立する。もちろん言葉で捉えられることだけが全てではありませんが、議論の場では言葉を尽くして“わからないもの”を見つけ、考え続ける姿勢がとても重要なのです」
 ブラインド鑑賞後、実際の画像をスクリーンで見た参加者たちはみな苦笑い。
ブラインド鑑賞後、実際の画像をスクリーンで見た参加者たちはみな苦笑い。
この日の最後には、「ワールドカフェ」と題したディスカッションの時間が設けられた。バイオテクノロジーをめぐる倫理上の課題や「生命とは何か」という問いをテーマに、1時間ほどかけて参加者全員で幅広く議論を行った。
ワールドカフェでの議論は、ルールに沿って進められる。まず4〜5人のグループに分かれ、簡単な自己紹介後、語り合うテーマを決めて15分間、議論する。制限時間が来たら、テーブルに残る1名のホストを決め、他のメンバーは別のテーブルに移動。ホストは、新しいグループメンバーに自分のテーブルで話し合われた内容を共有し、新たに議論を始める。このようなグループディスカッションを計4セット行うというものだ。結論を出すのではなく、あくまでも自由に意見を交換し、語り合うことを目的としている。
筆者が参加したテーブルでは、「DIYバイオの裾野が広がったとき、現状の生命倫理のルールは果たして機能するのか」「生命に対する科学的視点と情動的視点をどう融合させるのか」「宗教的・文化的な生命観の違いを生命倫理のルールづくりにどう反映させるのか」といったテーマで語り合った。
 テーブル上に用意された紙に思いつくまま議論の内容を書き連ねていく。
テーブル上に用意された紙に思いつくまま議論の内容を書き連ねていく。
とくに興味深かったのは、日本・韓国・ブラジルという異なるバックグラウンドを持つメンバーと「私たちの生命はいつから始まるのか」について議論したときだ。生物学的には、受精を起点に考えるのが合理的だろう。一方で、日本における法的な解釈で言えば、受精卵や胎児は「人間」とは見なされず(人の生命の萌芽などと呼ばれる)、出生の瞬間から人権が発生する。しかし、情動的な意味での“命の始まり”については、それぞれの宗教観が色濃く影響を与えている。例えばキリスト教のカトリックには「人は受精の瞬間から人間として尊重されるべき」という考え方があり、仏教には「生命という大きな流れの中で人は転生を繰り返す」という思想(輪廻転生)がある。そう考えると、地球上に最初の細胞が発生してから途切れなく紡がれてきた生命の営みの中で、固有のゲノムを持ち、他の誰とも異なる独自の存在として生きている私たちの命の始まりとは、いったいどこにあるのだろうか?
そんな問いに対して、ブラジル出身で筑波大学外国人受託研究員を務めるジウ・ヴィセンテ氏は「そもそも生命という概念はフィクションではないか」という視点を投げかけた。私たち人類は、壮大な「生命という名のフィクション」を、祈りや弔いといった宗教的行為によって補強しながら、現実のものとして信じているだけかもしれない。前提を解体し、生命に対する視座を拡張するジウ氏の指摘は、非常に印象的だった。
 ジウ・ヴィンセンテ氏(後列右)。
ジウ・ヴィンセンテ氏(後列右)。
議論は尽きず、願わくばもっと個別のテーマを深掘りする時間が欲しかったが、1時間で「ワールドカフェ」は終了。続いて、翌日のグループワークに備えたチーム分けに移った。参加者はまず、それぞれ関心のあるトピック3点と得意なこと(スキルセット)3点を紙に書き出す。その後、紙を掲げながら会場を歩き回り、関心領域が近い相手を探して4名1組のチームをつくる。筆者も3名のメンバーとチームを組むことができ、2日目のプログラムは終了した。
工学的に生命を追求するバイオインフォマティクスから生命をめぐる倫理上の課題まで、振れ幅の大きいテーマに向き合った2日目のワークショップ。この日の最大の学びは、「バイオテクノロジーの進化が突きつける課題は私たち自身の問題である」という当事者意識の芽生えだったように思う。今この瞬間も紡がれ続ける生命の営みの中で、私たち自身の生命性を揺さぶる新しい技術を、どのように実装していくのか。それは技術を操る主体であり、対象そのものでもある私たちが考え続けなければならない命題として、常に眼前に横たわっている。
「生命とは何か」。自らの生命性に思いを馳せる時、その目線はいつしか太古から連綿とつながる命の連鎖へと収斂していく。そして、その問いの矛先が、自らにも向かっていることに気づくのだ。今日はそんなメビウスの輪を生きるための作法を、少しだけ身につけた気がした。

バイオテクノロジーのさまざまな分野での応用の可能性について、参加者によるグループワークやプレゼンテーションを通じて議論します。バイオテクノロジーのさまざまな分野での応用の可能性について、参加者によるグループワークやプレゼンテーションを通じて議論します。
レポートを読む